FreeSurfer教程 #N. 实践教程之CookBook
- 实践#1:如何将一个个体subject映射到fsaverage?
- 实践2:如何提取stats文件夹内的统计信息?
- 实践#3:如何提取感兴趣ROI区域的结构信息?
- 实践#4:如果构建一个surface ROI重采样到体积(Volume)?
- 实践#5:如何使用FreeSurfer去除颅骨(skull-stripping)?
实践#1:如何将一个个体subject映射到fsaverage?
如何按自定义模板,重建皮层,并提取皮层信息?
实践2:如何提取stats文件夹内的统计信息?
- 方法: 使用
asegstats2table和aparcstats2table命令来提取
$ asegstats2table --subject <> --common-segs --meas <volume,mean,std> --stats=<stats file> --table=<extracted measurement to a text file>
$ asegstats2table --subjects sub-101 sub-103 --common-segs --meas volume --stats=aseg.stats --table=segstats.txt
--subjects选项指定了一个被试名称的列表。
--common-segs表示输出所有被试共有的分段,换句话说,如果一个受试者的分段数与其他受试者不同,不要以错误退出命令。
--meas表示要从表中提取哪种结构测量值(“volume “是默认值;替代值是 “mean “和 “std”)。
--stats指的是将从分段数据中提取的统计文件;
--table将提取的测量数据写入一个文本文件,按被试名称组织。
同理,aparcstats2table也类似,
$ aparcstats2table --subjects sub-101 sub-103 --hemi lh --meas thickness --parc=aparc --tablefile=aparc.txt
--hemi,指定要分析的半球
--meas,要提取的测量值,选项有"thickness”, “volume”, “area”, “meancurv”
--parc,指定图谱,选项有Desikan-Killinay图谱(“aparc”)和Destrieux图谱(“aparc.a2009s”)
实践#3:如何提取感兴趣ROI区域的结构信息?
- 如何将一个Volumetric ROI重采样到表面,然后从该ROI中提取结构测量值。
- 如何将ROI区域映射到皮层上
#!/bin/tcsh
setenv SUBJECTS_DIR `pwd`
# 使用AFNI的3dUndump创建5mm的ROI球体;ROI_file.txt 包含球心的 x、y 和 z 坐标
# https://afni.nimh.nih.gov/pub/dist/doc/program_help/3dUndump.html
3dUndump -srad 5 -prefix S2.nii -master MNI_caez*+tlrc.HEAD -orient LPI -xyz ROI_file.txt
# 使用tkmedit查看
# https://surfer.nmr.mgh.harvard.edu/fswiki/FsTutorial/TkmeditGeneralUsage
tkmedit -f MNI_caez_N27.nii -overlay S2.nii -fthresh 0.5
# 将结构像模板配准到fsaverage
fslregister --s fsaverage --mov MNI_caez_N27.nii --reg tmp.dat
# 在fsaverage上查看ROI
tkmedit fsaverage T1.mgz -overlay S2.nii -overlay-reg tmp.dat -fthresh 0.5 -surface lh.white -aux-surface rh.white
#将ROI映射到fsaverage皮层;
#https://surfer.nmr.mgh.harvard.edu/fswiki/mri_vol2surf
mri_vol2surf --mov S2.nii \
--reg tmp.dat \
--projdist-max 0 1 0.1 \
--interp nearest \
--hemi lh \
--out lh.fsaverage.S2.mgh \
--noreshape
# 检查ROI映射到膨胀皮层的情况
# https://surfer.nmr.mgh.harvard.edu/fswiki/tksurfer
tksurfer fsaverage lh inflated -overlay lh.fsaverage.S2.mgh -fthresh 0.5
实践#4:如果构建一个surface ROI重采样到体积(Volume)?
-
将一个由FreeSurfer创建的ROI投射到个体体积空间。
-
根据label信息,生成ROI Volume。
# 手动创建registration file(register.dat) # https://surfer.nmr.mgh.harvard.edu/fswiki/FsTutorial/ManualRegistration # 去掉--noedit参数,可以弹出GUI界面来手动调整;“beta_0001.nii” is a beta map created in the subject’s native space tkregister2 --mov beta_0001.nii --s subject --noedit --regheader --reg register.dat # 使用mri_label2vol命令将surface ROI转到体积空间。 # mri_label2vol: creates mgz volume from a label or set of labels # --temp: Template volume # --fillthresh: Relative threshold which the number hits in a voxel must exceed for the voxel to be considered a candidate for membership in the label. (See mri_label2vol --help for more information) # --proj: Project the label along the surface normal # https://surfer.nmr.mgh.harvard.edu/fswiki/mri_label2vol mri_label2vol --label lh.superiortemporal.label --temp beta_0001.nii --subject subject --hemi lh --fillthresh .9 --proj frac 0 1 .1 --reg register.dat --o $PWD/stgnew.nii -
mri_label2vol运行完成之后,我们得到一个新文件stgnew.ni,它是转换为体积空间的表面ROI。
实践#5:如何使用FreeSurfer去除颅骨(skull-stripping)?
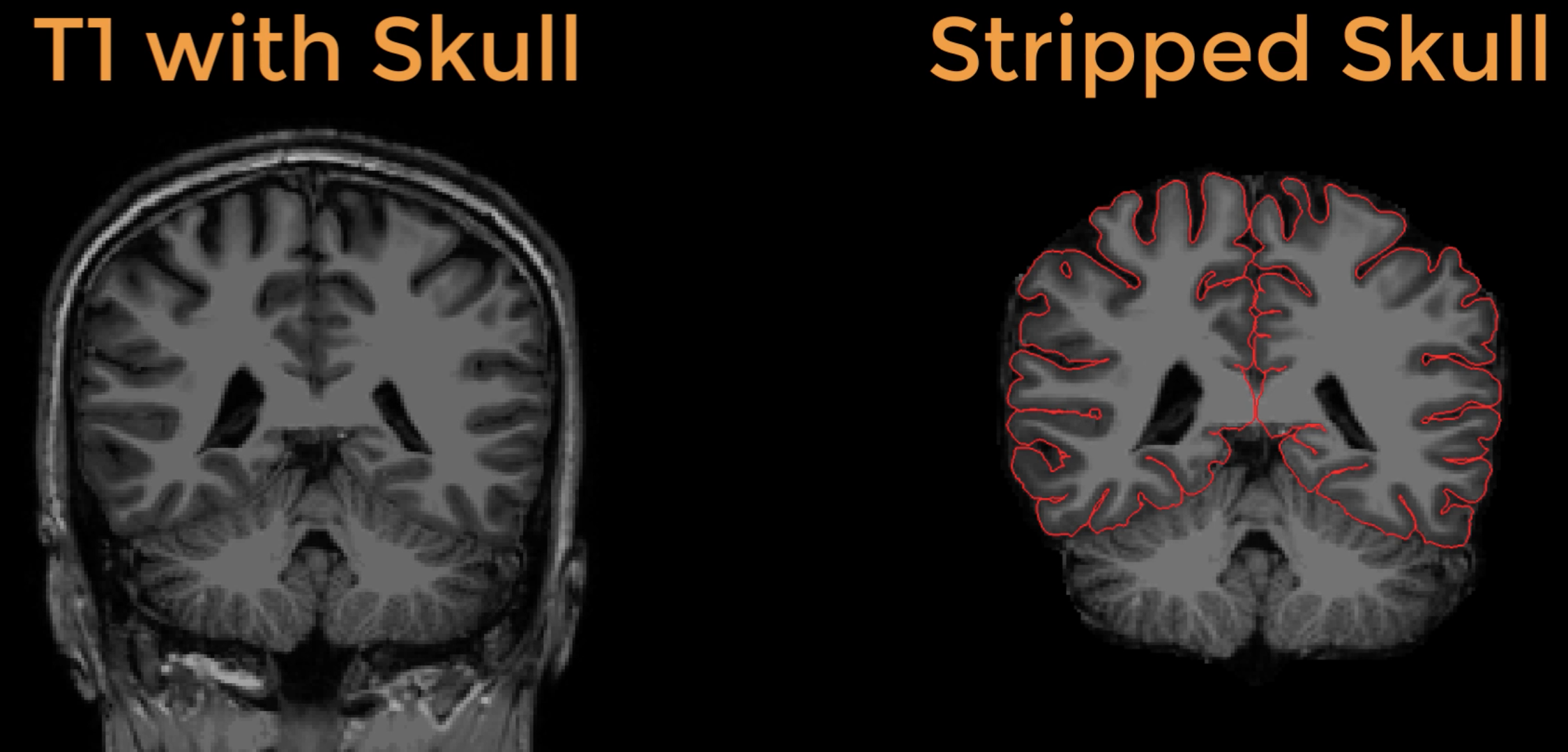
# 通过设置较低的分水岭阈值(例如 5)来去除更多的头骨,会生成颅骨去除的掩码文件brainmask.mgz。
recon-all -skullstrip -wsthresh 5 -clean-bm -s sub-117_ses-BL_T1w
# 即使分水岭阈值较低,仍有一些头骨(skull)和硬脑膜(dura)的碎片残留。你可以使用-gcut选项来删除后者
recon-all -skullstrip -clean-bm -gcut -subjid sub-117_ses-BL_T1w
在你使用watershed的gcut选项后,需要用以下代码重新生成皮层表面。
recon-all -autorecon2-pial -subjid <subject name>
参数解析:
- -subjid subjid:the subject data upon which to operate
- autorecon2-pial:process stages 21-23
- https://surfer.nmr.mgh.harvard.edu/fswiki/recon-all
扩展 可以使用FSL下的bet2工具来完成颅骨去除(很常用)。