FreeSurfer教程 #2. FreeSurfer输出结果与FreeView可视化
0. 本文知识点汇总
- FreeSurfer输出结果的简单理解
- 使用FreeView对FreeSurfer的结果(Volumes、Surfaces)进行可视化查看
1. 教程数据准备
在FsTutorial_Data下载相关教程的测试数据,大概8GB。
使用命令行下载相关教程数据,也可以直接访问网址下载,然后解压。
curl https://surfer.nmr.mgh.harvard.edu/pub/data/tutorial_data.tar.gz -o tutorial_data.tar.gz
tar -xzvf tutorial_data.tar.gz
rm tutorial_data.tar.gz
export TUTORIAL_DATA=/path/to/your/tutorial/dir # 定义环境变量TUTORIAL_DATA
ls $TUTORIAL_DATA
buckner_data fsfast-functional
diffusion_recons fsfast-tutorial.subjects
diffusion_tutorial long-tutorial
FreeSurfer 的输出结果
surf 文件夹下生成 . white、. sphere、. inflated 等网格点文件,每一个文件里面都存储了大脑皮质表面网格点的三维坐标及相邻顶点构成的三角面片信息。
surf 文件夹下生成基于曲面的形态特征数据,不同的特征采用不同的文件后缀名,
- 皮质厚度( . thickness )
- 雅可比度量(. jacobian. white)
- 脑沟( . sulc )
- 曲率(. curv)
- 外表面积(. area)
- 体积(. volume)等面数据文件,其坐标索引号与 Mesh 网格序号一致。
**stats **文件夹下,对于每个脑图谱(atlas)都有一个分区结果(parcellations)。比如,
-
lh.aparc.annot,使用Desikan-Killiany图谱的左半球的分区结果; -
lh.aparc.a2009s.annot,使用Destrieux图谱的左半球分区结果; -
aseg.stats包含了所有图谱的分割结果。如果想知道怎么提取这些信息,请跳转。
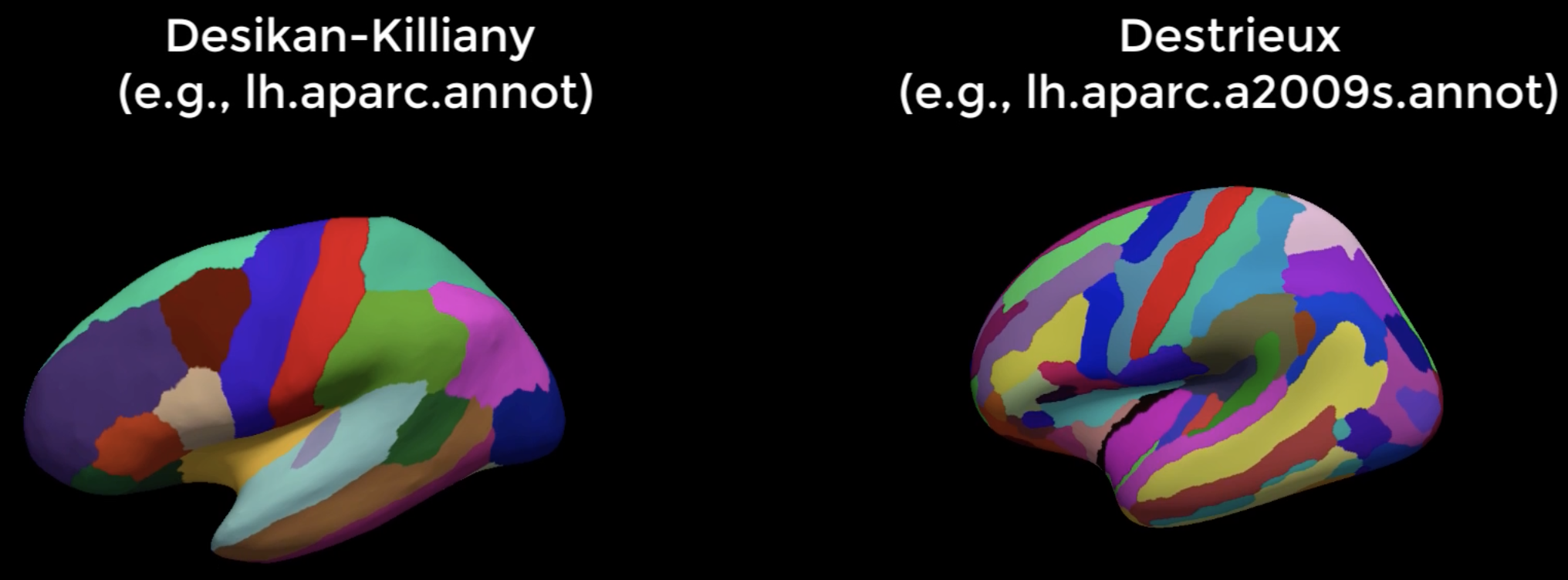
- Desikan-Killiany图谱与Destrieux图谱的区别在于,Destrieux图谱包含更多的分区,可以适用于更加精细化的分析。
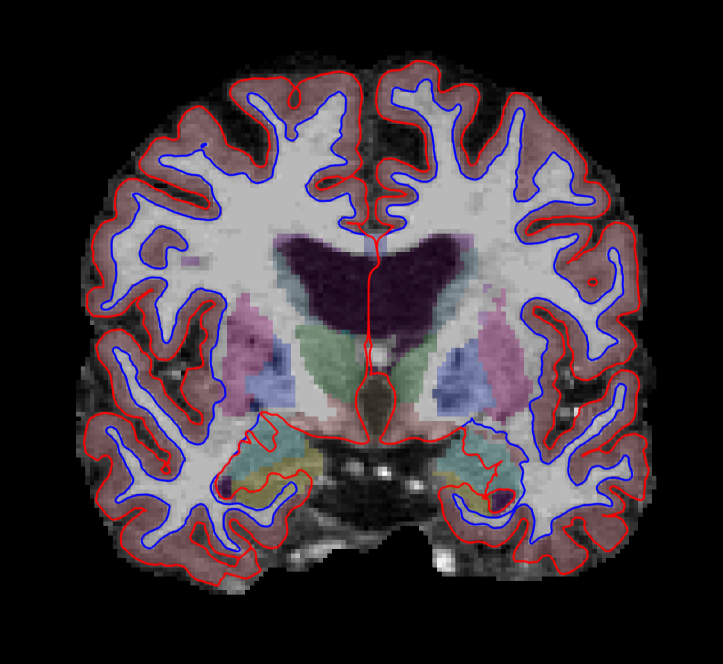
使用FreeView来图片查看Volumes
freeview -v \
good_output/mri/T1.mgz \
good_output/mri/wm.mgz \
good_output/mri/brainmask.mgz \
good_output/mri/aseg.mgz:colormap=lut:opacity=0.2 \
-f good_output/surf/lh.white:edgecolor=blue \
good_output/surf/lh.pial:edgecolor=red \
good_output/surf/rh.white:edgecolor=blue \
good_output/surf/rh.pial:edgecolor=red
-v用于加载volumes;-f用于加载surfaces;
使用FreeView来3D查看Surfaces
下述Surfaces都可以使用FreeView进行查看。
- pial(软膜), white(白质) and inflated surface(膨胀表面)
- sulcal(脑沟) and curvature maps(曲率图)
- thickness maps(厚度图)
- cortical parcellation(皮质分割)
freeview -f good_output/surf/lh.pial:annot=aparc.annot:name=pial_aparc:visible=0 \
good_output/surf/lh.pial:annot=aparc.a2009s.annot:name=pial_aparc_des:visible=0 \
good_output/surf/lh.inflated:overlay=lh.thickness:overlay_threshold=0.1,3::name=inflated_thickness:visible=0 \
good_output/surf/lh.inflated:visible=0 \
good_output/surf/lh.white:visible=0 \
good_output/surf/lh.pial \
--viewport 3d
参数以:+<cmd>=进行间隔区分,比如:annot=、:name=、:overlay=、:overlay_threshold=;
lh.pial:annot=aparc.annot表示在pial表面上加载Desikan-Killiany皮质分区;
lh.pial:annot=aparc.a2009s.annot表示在pial表面加载Destrieux皮质分区;
:name=pial_aparc:visible=0 表示更改显示名称并关闭该层显示;
lh.inflated:overlay=lh.thickness:overlay_threshold=0.1,3表示加载膨胀表面顶部的厚度叠加层并设置要显示的最小和最大阈值;
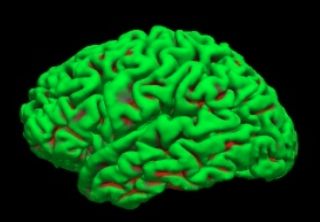
- 绿色区域是脑回,红色区域是脑沟
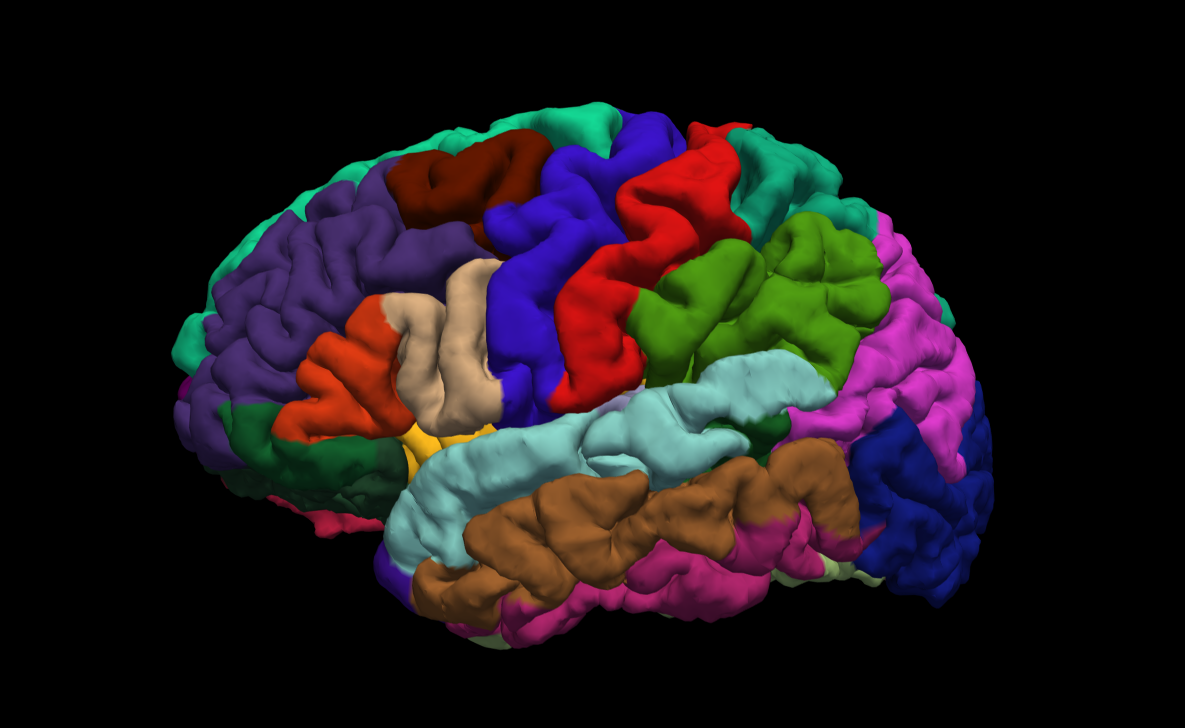
- 上图为Desikan-Killiany Cortical Parcellation
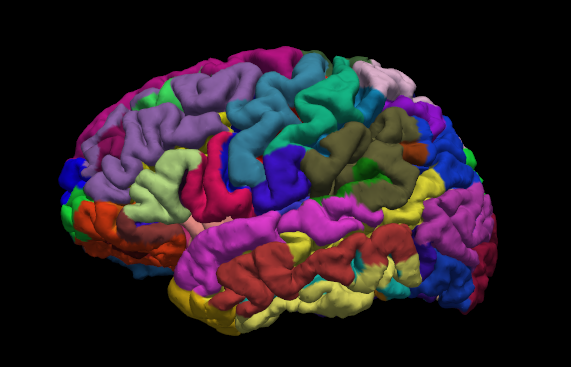
- 上图为Destrieux Cortical Parcellation
上述皮质分割可通过recon-all来生成,对应命令为:
?h.aparc.annot:
- Desikan-Killiany atlas
?h.aparc.a2009s.annot:
- Destrieux atlas
FreeView的简单使用方法,不需要每次在终端命令行输入命令来指定打开什么文件以及展示属性,而是仅输入freeview打开UI界面,点击操作即可,方便又快捷!
FreeSurfer 采用的是 RAS 坐标系,其意义为 R:right,X 轴正方向;A:anterior,Y 轴正方向;S:superior,Z 轴正方向。