AFNI 系列教程 #2.统计与建模
简介
现在,经过预处理后,我们可以对数据进行模型拟合。为了理解模型拟合的原理,我们需要回顾一些基本原理,如一般线性模型(GLM)、BOLD反应和什么是时间序列。这些主题在下面章节中都有讨论。
在你回顾了这些概念之后,你就可以使用FEAT进行一级分析了。下图说明了我们将如何对数据进行模型拟合。
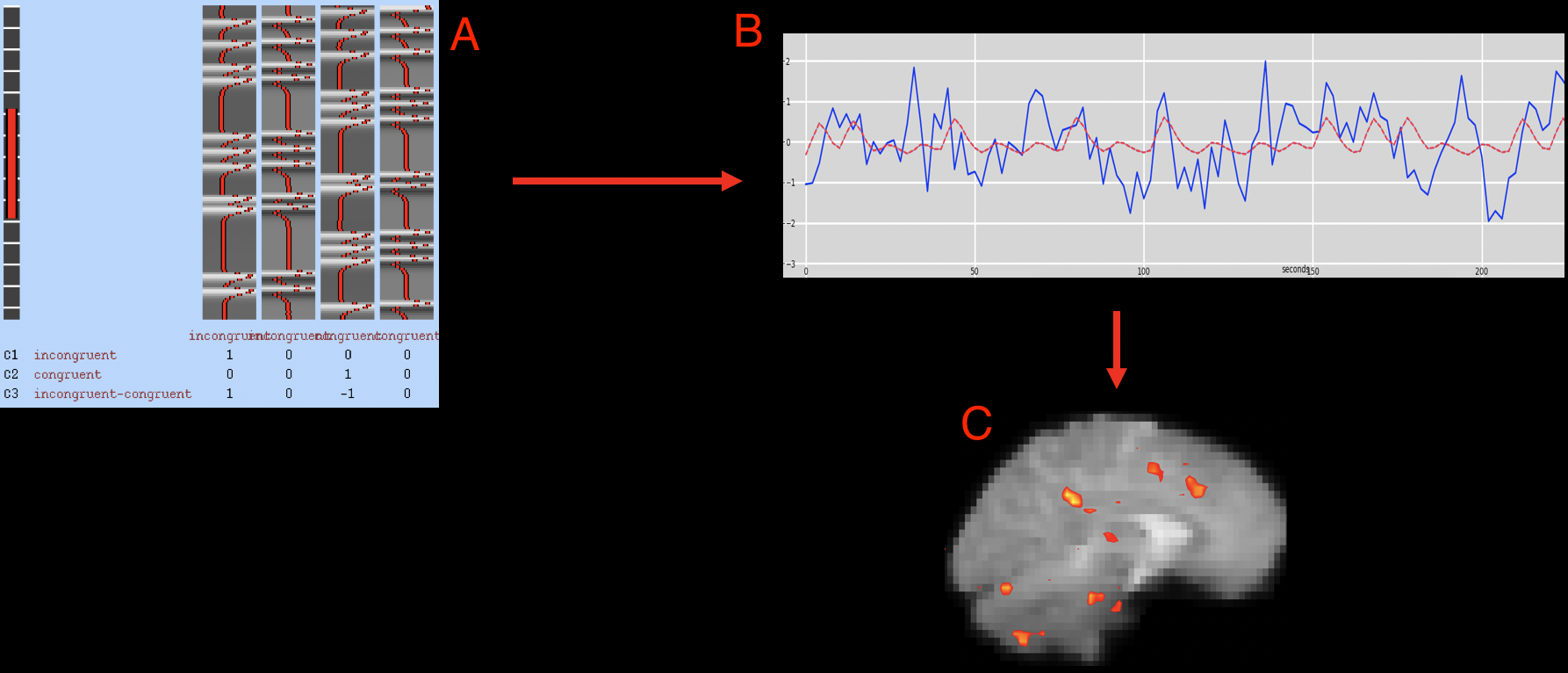
关于时间序列、BOLD信号、HRF、广义线性模型相关基础知识,可参阅FSL教程。
运行一级分析(First-Level Analysis)
uber_subject.py 脚本的再次使用
之前,我们使用uber_subject.py命令为单个研究对象设置了预处理脚本。你可能还记得,我们删除了其中一个名为"regress"的处理块,因为我们当时并不关注回归。但现在,我们将把回归块添加回uber_subject.py脚本中,并将预处理和一级分析合并到一个脚本中。
让我们为同一个被试sub-08创建一个新脚本。首先,导航到sub-08目录,输入rm -r subject_results,删除预处理目录。在包含所有被试的目录下,在命令行中输入uber_subject.py。这一次,在"analysis initialization"部分,我们将保留所有数据块的原样(不过,由于本示例数据集中的数据已经进行了切片时间校正,因此可以删除"tshift “数据块而不影响结果,这一点在前面的章节中已经讨论过)。
按照预处理的方法填写解剖和功能图像,并对 “extra align options"和 “extra tlrc options"进行同样的修改。本章我们将重点讨论图形用户界面的以下部分:
- stimulus timing files;
- symbolic GLTs;
- extra regress options.
如果uber_subject.py不能使用,比如pyqt4老旧安装麻烦等问题,导致脚本不能使用,建议使用下述使用uber_subject.py脚本输出的sub_08_afni_proc.sh脚本。
#!/usr/bin/env tcsh
# created by uber_subject.py: version 1.2 (April 5, 2018)
# creation date: Mon Nov 18 12:30:05 2019
# set subject and group identifiers
set subj = sub_08
set gname = Flanker
# set data directories
set top_dir = ${PWD}/sub-08
set anat_dir = $top_dir/anat
set epi_dir = $top_dir/func
set stim_dir = $top_dir/func
# run afni_proc.py to create a single subject processing script
afni_proc.py -subj_id $subj \
-script proc.$subj -scr_overwrite \
-blocks tshift align tlrc volreg blur mask scale regress \
-copy_anat $anat_dir/sub-08_T1w.nii.gz \
-dsets \
$epi_dir/sub-08_task-flanker_run-1_bold.nii.gz \
$epi_dir/sub-08_task-flanker_run-2_bold.nii.gz \
-tcat_remove_first_trs 0 \
-align_opts_aea -giant_move \
-tlrc_base MNI_avg152T1+tlrc \
-volreg_align_to MIN_OUTLIER \
-volreg_align_e2a \
-volreg_tlrc_warp \
-blur_size 4.0 \
-regress_stim_times \
$stim_dir/congruent.1D \
$stim_dir/incongruent.1D \
-regress_stim_labels \
congruent incongruent \
-regress_basis 'GAM' \
-regress_censor_motion 0.3 \
-regress_motion_per_run \
-regress_opts_3dD \
-jobs 8 \
-gltsym 'SYM: incongruent -congruent' -glt_label 1 \
incongruent-congruent \
-gltsym 'SYM: congruent -incongruent' -glt_label 2 \
congruent-incongruent \
-regress_reml_exec \
-regress_make_ideal_sum sum_ideal.1D \
-regress_est_blur_epits \
-regress_est_blur_errts \
-regress_run_clustsim no
刺激时序文件(Stimulus Timing Files)
我们的目标是创建拟合的时间序列,以便在组级分析中使用估计的贝塔权重。但要做到这一点,我们首先需要创建理想的时间序列。
让我们来看看Flanker数据集。在每个受试者的func目录中都有标为events.tsv的文件。这些文件包含我们创建timing files(也称为onset files)所需的三项信息:
-
条件名称;
-
相对于扫描开始,每次试验发生的时间(以秒为单位);
-
每次试验的持续时间。 这些信息需要从
events.tsv文件中提取,并以AFNI软件可以读取的方式格式化。在这种情况下,我们将为每个条件创建一个时序文件,然后根据条件所处的运行情况分割该文件。这样,我们总共将创建四个时序文件: -
第一次运行中出现的不一致试验的时序(我们称之为 incongruent_run1.txt);
-
第二次运行中出现的不一致试验的时序(incongruent_run2.txt);
-
在第一次运行过程中进行的一致试验的时序(congruent_run1.txt);
-
第二次运行中出现的一致试验的时序(congruent_run2.txt)。
每个时序文件的格式都相同,由三列组成,顺序如下:
- 开始时间,以秒为单位,相对于扫描开始时间;
- 试验持续时间,以秒为单位;
- 参数调制(Parametric modulation)。
每次run的时序文件将浓缩为每个条件的单个时序文件,分别称为incongruent.1D和congruent.1D。1D扩展名是 AFNI 特有的,表示文件包含按行和列排列的文本。
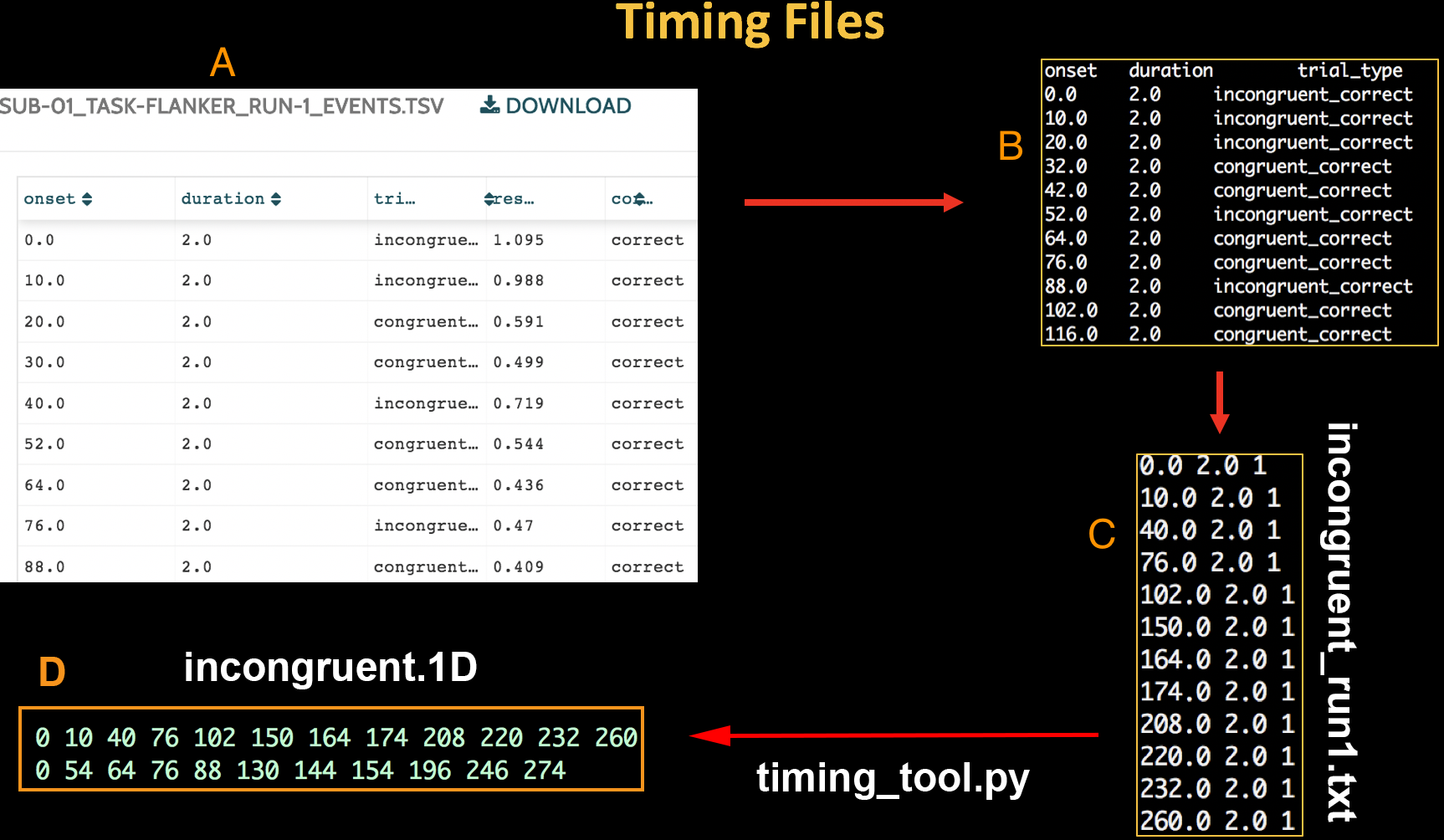
要格式化时序文件,请下载此脚本。(点击 Raw 按钮,然后在新打开的窗口中右击并选择 “另存为"即可下载)或者直接复制下述make_Timings.sh脚本。我们不会详细介绍它的工作原理,但你只需将其放在包含实验对象的实验文件夹中,然后输入bash make_Timings.sh。这将为每个实验对象的每次run创建时序文件,并将其存储在每个实验对象对应的func目录中。要检查输出,请键入 cat sub-08/func/incongruent.1D。你应该会看到与上图类似的数字。
#!/bin/bash
#Check whether the file subjList.txt exists; if not, create it
if [ ! -f subjList.txt ]; then
ls | grep ^sub- > subjList.txt
fi
#Loop over all subjects and format timing files into FSL format
for subj in `cat subjList.txt`; do
cd $subj/func
cat ${subj}_task-flanker_run-1_events.tsv | awk '{if ($3=="incongruent_correct") {print $1, $2, 1}}' > incongruent_run1.txt
cat ${subj}_task-flanker_run-1_events.tsv | awk '{if ($3=="congruent_correct") {print $1, $2, 1}}' > congruent_run1.txt
cat ${subj}_task-flanker_run-2_events.tsv | awk '{if ($3=="incongruent_correct") {print $1, $2, 1}}' > incongruent_run2.txt
cat ${subj}_task-flanker_run-2_events.tsv | awk '{if ($3=="congruent_correct") {print $1, $2, 1}}' > congruent_run2.txt
#Now convert to AFNI format
timing_tool.py -fsl_timing_files congruent*.txt -write_timing congruent.1D
timing_tool.py -fsl_timing_files incongruent*.txt -write_timing incongruent.1D
cd ../..
done
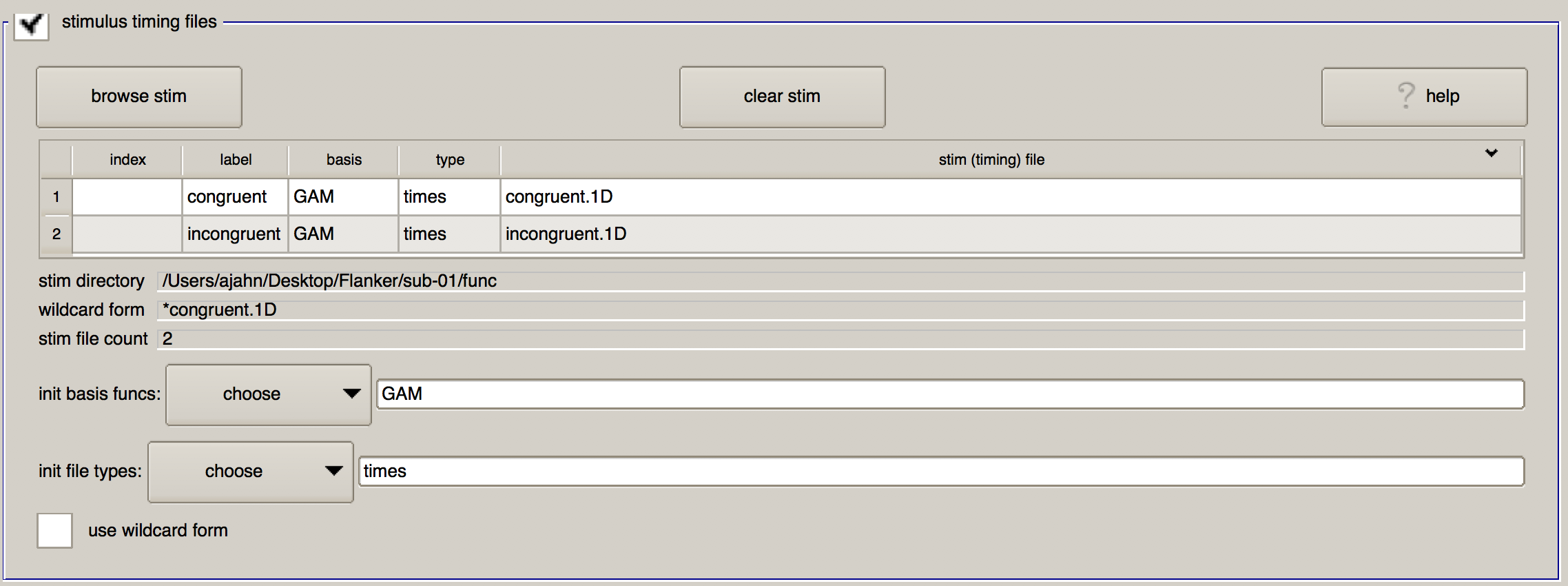
在上文创建了时序文件后,点击 “browse stim(浏览刺激)“按钮,选择位于sub-01/func 中的congruent.1D和incongruent.1D文件。(你可以按住命令键并点击单个文件来选择多个文件)。
单击 “OK(确定)“后,你将看到一个表格,其中包含你选择的时序文件。除了显示所选时序文件的名称外,还有另外三列,分别名为 “label”、“basis"和 “type”。“Label"是时序文件在进行模型拟合的命令(即3dDeconvolve)中的引用方式,而 “basis"和 “type"则指定了应用于时序文件的基础函数。
GAM的默认基函数规定,起始时间应与典型 HRF 进行卷积。这种基函数只需要估计一个参数,即HRF的高度,它大致相当于神经活动对该条件的反应量。你可以应用的其他基函数包括 BLOCK 函数(即 HRF 章节中讨论过的boxcar regressor)、TENT函数用于估计条件开始后指时序间点的活动和SPMG2包括时间导数。
下面的"init file types"字段允许你指定希望使用的卷积类型。默认的times表示对时序文件中指定的所有时间点进行卷积,并为 HRF 生成平均最适合所有发生情况的参数估计;而IM则会为每次试验估算单独的贝塔权重(可用于更高级的分析,如贝塔序列分析)。AM1和AM2选项用于参数调制分析(不在本教程范围内),而files则表示不应用卷积–这对运动等干扰回归因子很有用。
完成文件加载后,图形用户界面的这一部分应该如下所示:
广义线性测试(Symbolic GLTs)
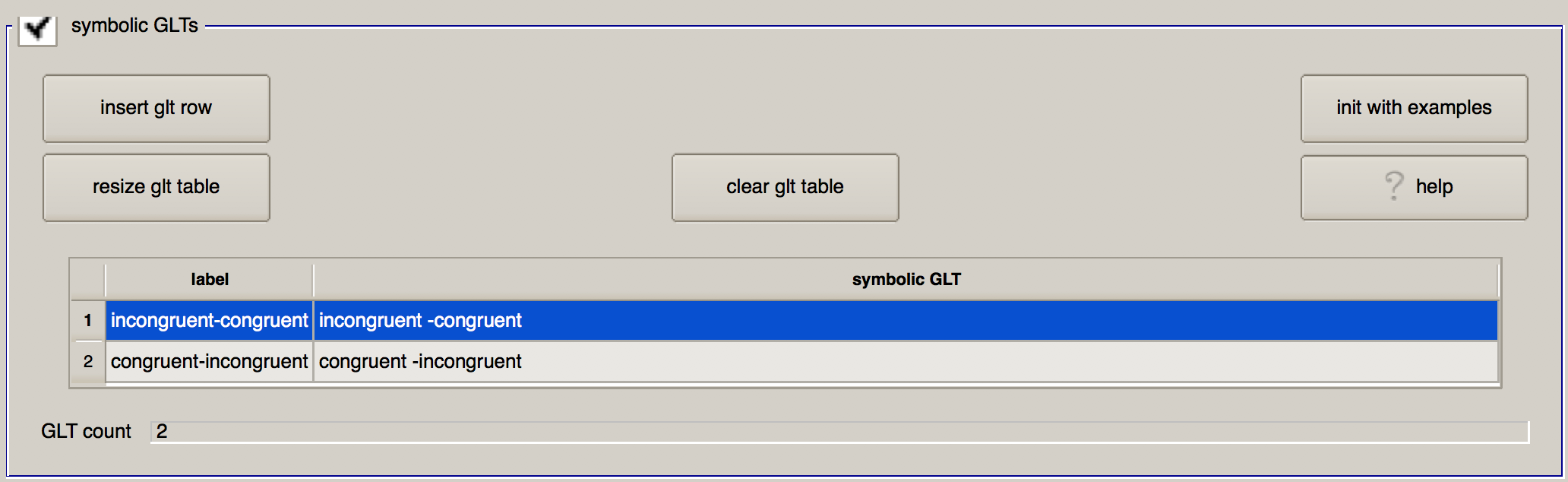
“symbolic GLT “允许你指定广义线性检验,这些检验将在根据上述条件估算出上述贝塔权重后进行计算。这里的符号有点奇怪,最简单的方法可能是点击"init with examples"按钮来显示设置检验的示例语法。点击后,你将看到两个对比: C-I 和 mean.CI。
让我们来看看第一个对比: 它使用对比度权重来指定如何计算不同条件之间的对比度,并在"刺激时序文件"部分指定的标签前加上对比度权重。**无符号意味着对比度权重为+1,而负号意味着对比度权重为-1。因此,“一致-不一致(congruent -incongruent)“这一行意味着 “一致"条件下的参数估计值权重为+1,而"不一致"条件下的参数估计值权重为-1,然后取两者之差。**使用不同的计算方法,第二个对比取两个条件的平均值,即两个条件都乘以0.5。
一般来说,计算不同条件间差异的对比权重总和应为0,而计算不同条件间平均值的对比权重总和应为1。
让我们修改这个表格来练习 GLT 语法,并设置我们想要的对比。在第一行中,将 “label"列改为 “不一致-一致(incongruent-congruent)",在 “symbolic GLT “列中输入"不一致-一致(incongruent-congruent)"。同样,在第二行中指定congruent -incongruent对比。完成后,该部分应如下所示:
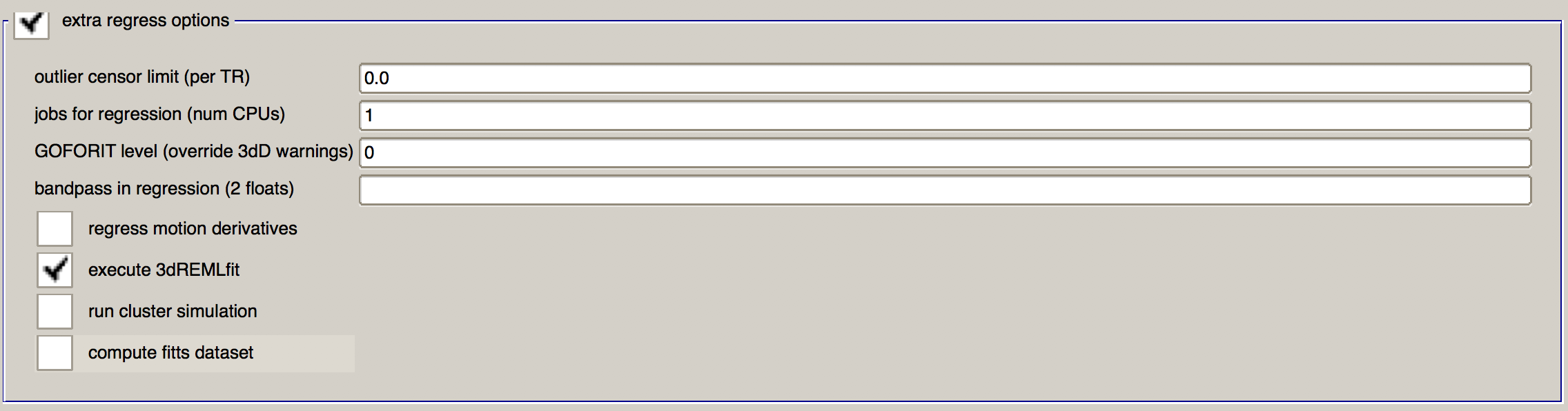
额外的回归选项(Extra Regress Options)
最后,我们将说明对3dDeconvolve命令的偏好。
第一个字段是 “离群值删减限制(outlier censor limit)",它将从分析中移除离群值删减分数大于右侧字段指定值的任何TR。(这些离群值是由3dToutcount检测到的,它会标记出 TR 中信号强度与脑掩膜中其他体素相比超过3个标准差的任何体素)。如果将该值设为 0.0,则不会根据3dToutcount的输出结果对任何 TR 进行删减。由于运动量大的体块会被剔除,因此我们暂时不做调整。
下一个字段 “用于回归的工作(CPU 数量),jobs for regression (num CPUs)“指定了用于回归分析的处理器数量。由于该步骤对计算要求较高,因此请使用最大 CPU 数量。在本例中,我将其设置为8。
“GOFORIT 级别(覆盖 3dD 警告),GOFORIT level (override 3dD warnings)“将忽略3dDeconvolve检测到的任何有关设计矩阵的警告。一般来说,除非确定矩阵误差可以忽略不计,否则不应使用 GOFORIT。一般情况下,当3dDeconvolve检测到两个或多个回归因子之间存在异常高的共线性时,就会发出警告并停止运行。
“bandpass in regression"字段通常用于静息态分析,以去除低频和高频波动。 但对于任务数据,低通滤波(即去除高频信号)有可能会去除与任务相关的实际信号。此项留空。
此外还有四个复选框。“回归运动导数(Regress motion derivatives)“将对运动回归器的高阶导数进行建模,从而捕捉到更复杂的头部运动。这对于运动频繁的人群非常有用,例如儿童或某些临床受试者;而且只要你的数据时间序列较长(例如一次运行中超过 200 个 TR),估计这些额外参数的自由度可能不会用完。在本教程中,我将不选中该选项,但你也可以自行决定。
我也没有选中"run cluster simulation"复选框,因为它会在你改变阈值滑块时实时计算聚类是否具有统计意义。由于我一般不对单个受试者进行推断–我们稍后会在群体水平上进行推断–所以我省略了这个选项。不过,我还是选中了 “execute REMLfit “选项,因为这将创建一个单独的统计数据集,与传统的3dDeconvolve方法相比,能更好地考虑时间自相关性。稍后,我们可以使用3dREMLfit的输出来使用受试者参数估计的可变性信息,以创建更精确的组级推断图。
完成后,这一部分应该是这样的:
现在,依次点击最上面的三个图标(纸片、放大镜和绿色的 “开始"按钮)来运行预处理和回归。总共大约需要 5-10 分钟。
理想时间序列与广义线性模型
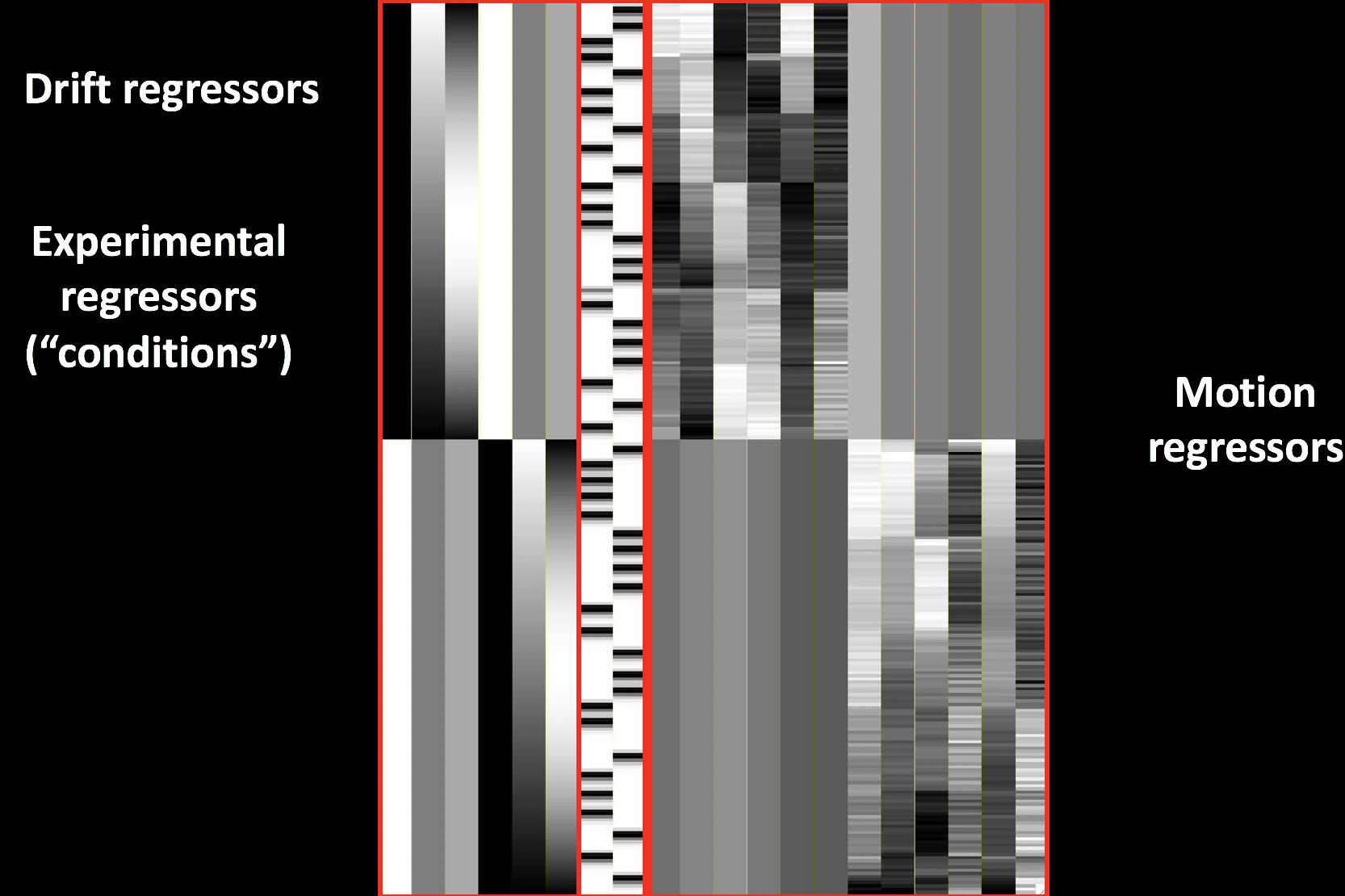
在等待分析完成的同时,让我们来看看刚刚创建的模型与 GLM 的关系。请记住,每个体素都有一个 BOLD 时间序列(我们的结果测量),我们用 Y 表示。这些回归因子构成了我们的设计矩阵,我们用一个大的 X 来表示。
到目前为止,所有这些变量都是已知的–Y 是通过数据测得的,而 x1 和 x2 则是通过卷积 HRF 和时序起始值得到的。由于矩阵代数用于设置设计矩阵和估算贝塔权重,因此方向要转 90 度: 通常,我们认为时间轴是从左至右,但现在我们将其描述为从上到下。换句话说,运行的起始点位于时间轴的顶端。
GLM 方程的下一部分是贝塔权重,我们用 B1 和 B2 表示。这表示我们对每个回归因子需要缩放的 HRF 的估计值,以便与 Y 中的原始数据最匹配–因此称为 “贝塔权重”。等式中的最后一项是 E,表示残差,即我们理想的时间序列模型与估计贝塔权重后的数据之间的差异。如果模型拟合得好,残差就会减少,而且一个或多个贝塔权重更有可能具有统计意义。
检查输出结果
当脚本完成后,导航到文件夹sub-08/subject_results/group.Flanker/subj.sub08/sub08.results。除了之前看到的预处理块,你还将看到统计数据集: 标有stats.sub_08+tlrc的数据集已使用传统的3dDeconvolve方法进行了分析;数据集stats.sub_08_REML+tlrc已考虑了时间自相关性。
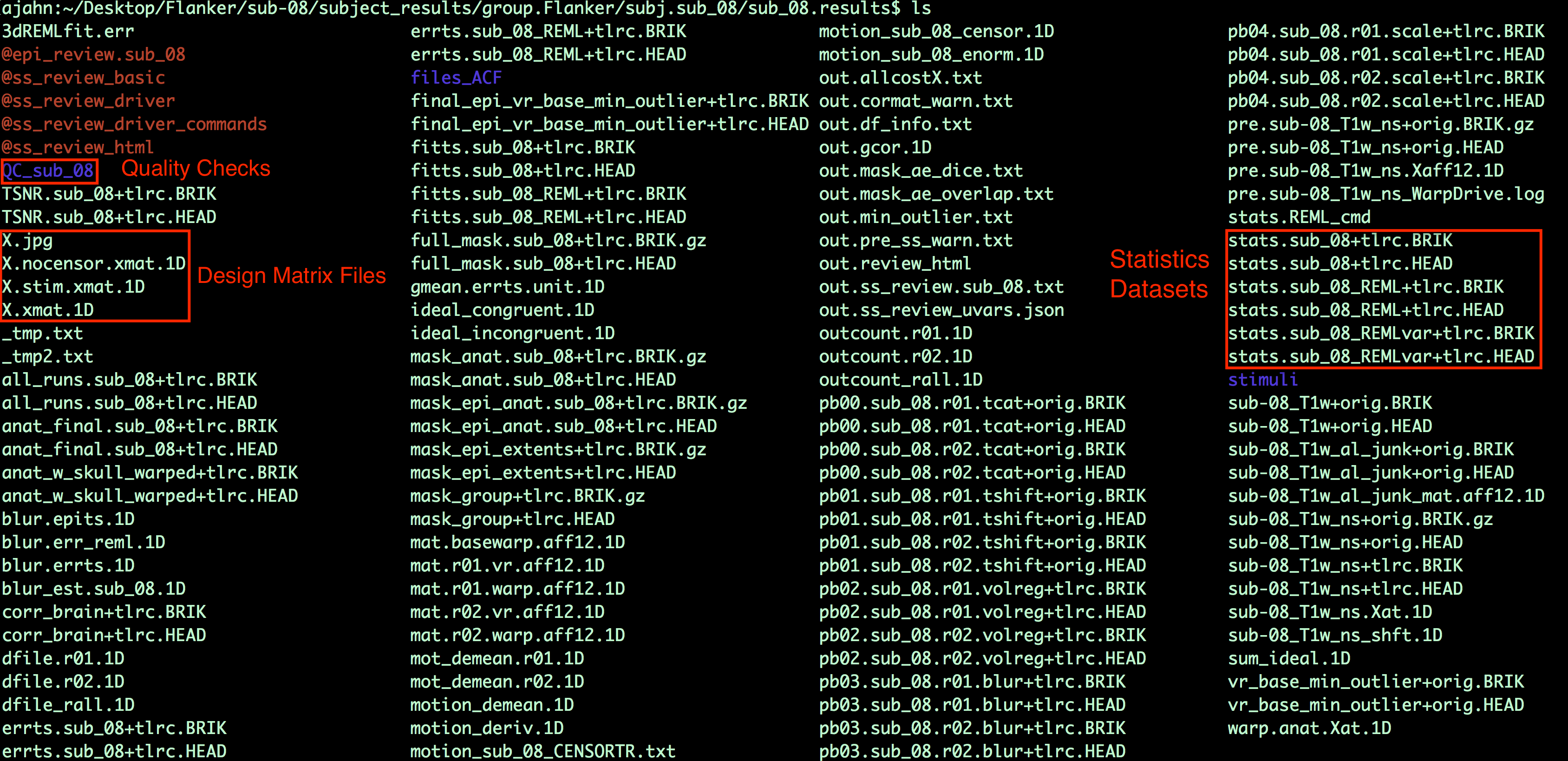
你还会看到一些以 “X “开头的文件,如X.xmat.1D。这些文件代表设计矩阵的不同部分。例如,你可以输入aiv X.jpg查看设计矩阵:
如果要查看不同的视图,在单独的行中查看所有回归因子,请键入1dplot -sepscl X.xmat.1D:
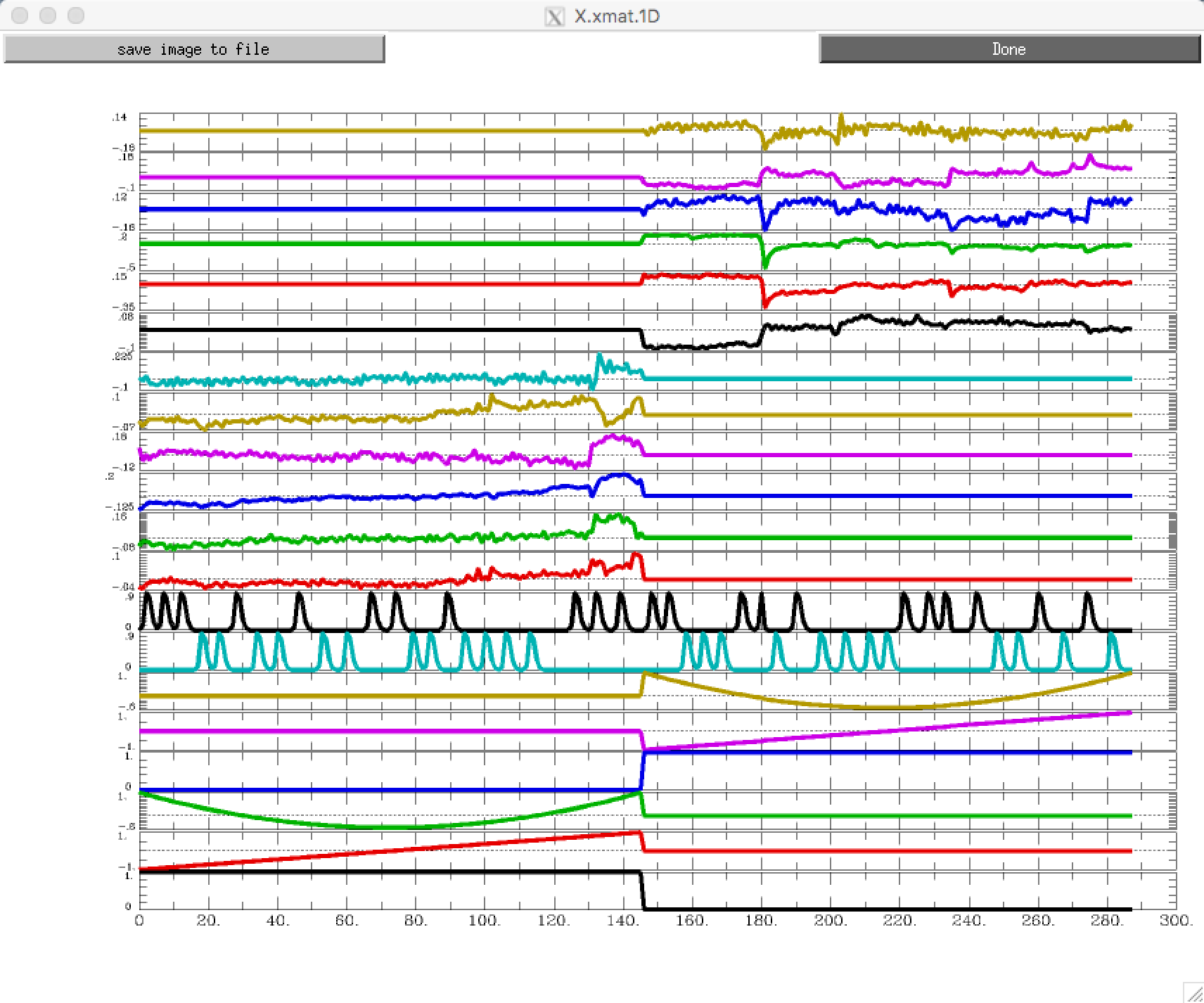
确保设计矩阵看起来合理。低频漂移的建模是否符合你的预期?每个条件下试验的起始时间是否与你在上一章中创建的定时文件一致?
查看统计文件
现在我们可以查看数据的对比图了。输入afni打开图形用户界面,选择anat_file.sub_08作为底图。(如果你已将 MNI152 模板复制到当前目录,或将其放在 aglobal 目录中,也可以使用该模板)。选择stats.sub_08作为覆盖层。你应该会看到如下内容
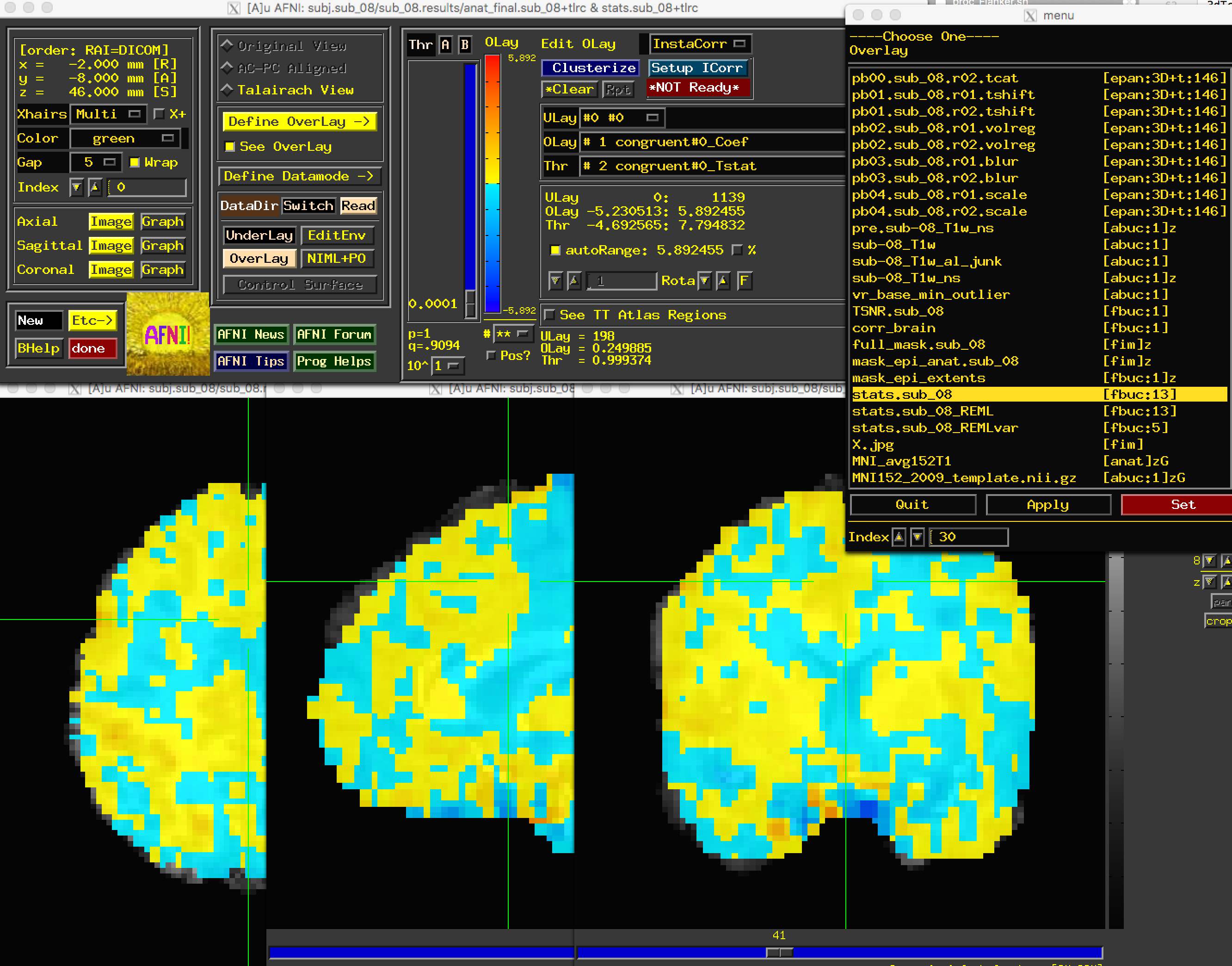
虽然 AFNI 查看器看起来有太多的选项,但一旦你对它们越来越熟悉,就可以自定义查看结果的方式。如果这是你第一次在 AFNI 中查看统计数据,那么 “Define Overlay"面板中最引人注目的功能将是滑块栏(slider bar)(允许你对图像进行阈值设置,使其只显示高于某一数值的值),以及 “ULay”、“OLay"和 “Thr"菜单,它们分别与 “Underlay”、“Overlay “和 “Threshold “子框相对应。
让我们从滑动条开始。如果上下移动它,你会看到体素要么消失,要么重新出现。这是因为我们正在阈值化或移除低于滑动条左侧阈值数字的体素。这个数字将基于在 “Thr"菜单中选择的子块;在本例中,我们打开查看器时选择的子块是第2个volume,即 “一致性"条件下贝塔权重的 T 统计图。当你将滑块移动到例如 1.9753 的值时,你还会注意到滑块条下方的数字p=也发生了变化,变成了0.493。这表示当前所选阈值图的未校正 p 值阈值;换句话说,任何彩色体素都通过了 0.493 的单个 p 值阈值。
如果你想设置特定的未校正 p 值阈值,请右键单击p= 文本,选择 “set p-threshold”,然后键入你想要的阈值(例如 0.001)。
当我们还没有多重比较校正时,因此我们无法判断任何一个体素是否具有统计学意义。不过,在未经校正的 p 值阈值下查看数据,可以让你对统计数据的空间布局有一个大致的了解,并表明结果的趋势是否与你预测的方向一致,或者是否出现了一些问题。例如,高度相关的回归因子会有非常大的参数估计值和相应的高变异性。你还应该确保,在相对较高的未校正 p 值阈值(例如 p=0.01 或更高)下,激活一般位于灰质内。例如,如果在脑室中发现大量 “活跃 “体素,则可能表明数据质量有问题。
现在,将 OLay 子项改为incongruent-congruent#0_Coef,将 Thr 子项改为incongruent-congruent#0_Tstat,并将未校正 p 值阈值设为 0.05。在大脑中点击,观察哪些地方的统计数据为正值,哪些地方为负值。你发现哪些地方有明显的激活体素 “集群”?它们在你预期的位置吗?
稍后,你将学习到一种名为簇校正(cluster correction)的多重校正技术。这种方法可以寻找由通过给定未校正阈值的体素组成的簇,然后确定该簇是否重要。在本章中,我们将不讨论如何计算聚类需要多大,但现在可以点击 “*Clusterize"按钮,将体素数目改为 45。这样,你将只看到那些由 45 个或更多体素组成的聚类,且每个体素都通过了 0.05 的未校正 p 值阈值。你可以点击 “Rpt(报告)“按钮,查看通过该阈值的每个群集的报告,其中列出了体素大小、群集峰值体素的位置,以及将十字准线移动到群集并使其闪烁的选项。
练习
-
在 “Define Overlay"面板的右下角,你会看到写有
OLay =和Thr =的文字,以及等号右侧的一些数字。这些数字表示交叉线所在体素的叠加和阈值数据集的值;换句话说,就是参数(或对比度)估计值以及相关的 t 统计量。选择 4 号子区间(incongruent#0_Coef)作为叠加,并写下 OLay = 字段中显示的相应数字。对子区间 #1(全等#0_Coef)做同样的操作。现在选择 7 号子模块(incongruent-congruent_GLT#0_Coef),但在此之前,请想一想你期望的值是什么。现在选择子项。它与你预测的相符吗?为什么或为什么不一致? -
执行练习 1 中的相同步骤,查看子边框 #7 和 #10 的对比。你发现叠加值有什么变化?从对比度权重来看,这合理吗?
-
你可以使用图像计算器计算贝塔权重之间的差值。每个主要软件包都有一个;AFNI 的计算器称为
3dcalc。在 sub-08.results 中输入3dcalc -a stats.sub-08+tlrc'[4]'-b stats.sub-08+tlrc'[1]'-expr'a-b'-prefix Inc-Con+tlrc,即可计算出 sub-08 的不一致和一致贝塔权重之差。阅读这条命令的方法是,它将统计数据集的第 4 个子数据集分配给变量 “a”,将统计数据集的第 1 个子数据集分配给变量 “b”。我们知道这两个变量分别对应于不一致和一致的贝塔权重,然后通过在 -expr 选项中输入数学表达式 “a-b"来进行对比。然后,我们选择一个前缀,在本例中为 “Inc-Con”。结果输出应该与使用 Inc-Con 作为叠加时在结果查看器中看到的结果相同。现在用同样的方法创建一个名为 “Con-Inc “的输出文件,将第 1 个子条带与第 4 个子条带进行对比。 -
虽然默认的配色方案和显示效果足以满足大多数用途,但你可能还是想根据自己的喜好和审美进行更改。例如,一旦你设置好统计叠加的阈值,请单击 AFNI GUI 中的 “Define Datamode"按钮,单击 “Warp Olay on Demand”,并为
OLay Resam mode选择Cu重采样方法(“Cubic"插值)。这将平滑在你使用的阈值下存活的体素边缘。回到 “Define Overlay"面板,右键单击颜色栏,选择不同的调色板,看看哪种最合适。