AFNI 系列教程 #1.Preprocessing
简介
我们在对fMRI数据进行预处理时,会对每个TR获取的三维图像进行清理。一个fMRI容积(volume)不仅包含我们感兴趣的信号–含氧血液的变化,还包含我们不感兴趣的波动(fluctuations),如头部运动(head motion)、随机漂移(random drifts)、呼吸(breathing)和心跳(heartbeats)。我们将这些波动称为噪声(noises),因为我们希望将它们从我们感兴趣的信号中分离出来。其中一些可以通过建模从数据中回归出来(将在建模拟合一章中讨论),其他的可以通过预处理减少或去除。
数据前提:请提前准备好Flanker task数据集,我们可以在OpenNeuro网站上找到并免费下载。
要开始预处理sub-08的数据,请通读以下章节。我们将首先概述如何使用AFNI命令,然后介绍uber_subject.py,它允许编写一个脚本,为我们完成所有预处理。然后,您将了解为什么要进行这些预处理步骤,以及如何在每个步骤之前和之后检查数据质量。
预处理步骤
不同的软件包会以略微不同的顺序完成这些步骤–例如,FSL会在模型拟合后对统计图(statistical map)进行归一化处理。还有一些分析省略了某些步骤–例如,一些做多体素模式分析(multi-voxel pattern analyses)的人不平滑他们的数据。无论如何,下面列出的是在典型数据集上执行的最常见步骤。
1. AFNI命令与uber_subjects.py
简介
在所有fMRI分析软件包中,AFNI有最难学的名声。尽管过去可能确实如此,但AFNI的开发人员在过去几年中努力使他们的软件更易学易用:除了查看器,AFNI的最新版本还包含其他图形用户界面,可以通过命令uber_subject.py和uber_ttest.py访问这些图形界面。这些图形用户界面用于创建脚本,自动完成每个被试的预处理和模型设置。
在讨论这些命令之前,我们先回顾一下典型AFNI命令的基本原理。毕竟,“uber"脚本只是将大量命令按照处理数据的顺序编译在一起。您还将使用单个AFNI命令执行更高级的分析,如感兴趣区域(ROI)分析。
AFNI 命令
AFNI命令类似于Unix命令: 它们通常需要至少一个参数或输入,而且通常还需要指定命令输出的名称。
以头骨剥离为例,这是一个常见的预处理步骤,用于将头骨从大脑中剥离出来。执行这一步的AFNI命令称为3dSkullStrip。导航至sub-08/anat目录,然后输入3dSkullStrip并按回车键,我们可以看到该命令的帮助文档信息。
通常情况下,只键入命令而不输入任何参数将默认打印帮助文档。在这里我们需要指定一个额外的标志-h来打印帮助文件,输入3dSkullStrip -h然后按回车键。你会注意到屏幕上打印了大量的文本,超过了终端可以同时显示的数量。如果想看更容易阅读的帮助文件,键入3dSkullStrip -h | less。竖条表示竖条左边命令的输出,即"3dSkullStrip -h”,应该被导入less命令,它允许你上下翻阅帮助文件。在这个 “分页窗口"中,输入"d"可以向下翻一页,输入"u"可以向上翻一页,输入上下箭头可以上下翻一行。要搜索帮助文件,键入一个正斜线(/),然后键入要查找的文本,按回车键。要退出分页窗口,请按 “q"键。
**文档和帮助文件是AFNI的最大优势。每条命令的用法都有清晰的概述,并详细解释了使用不同选项的原因。**给出的示例命令涵盖了不同的情况–例如,如果颅骨去除后在输出图像中留下了太多的颅骨,我们建议使用-push_to_edge这样的选项。
3dSkullStrip最基本的用法是使用-input标志来指定将要被剥离的解剖数据集。例如
3dSkullStrip -input sub-08_T1w.nii.gz
大约一分钟后,会生成一个名为skull_strip_out+orig的新文件。这就是头骨切片的解剖图像,我们可以打开AFNI浏览器查看。你可能会注意到在额叶有几个体素的皮质被移除,头骨顶部和后部有一些硬脑膜残留,但总体来说头骨剥离的效果非常好。
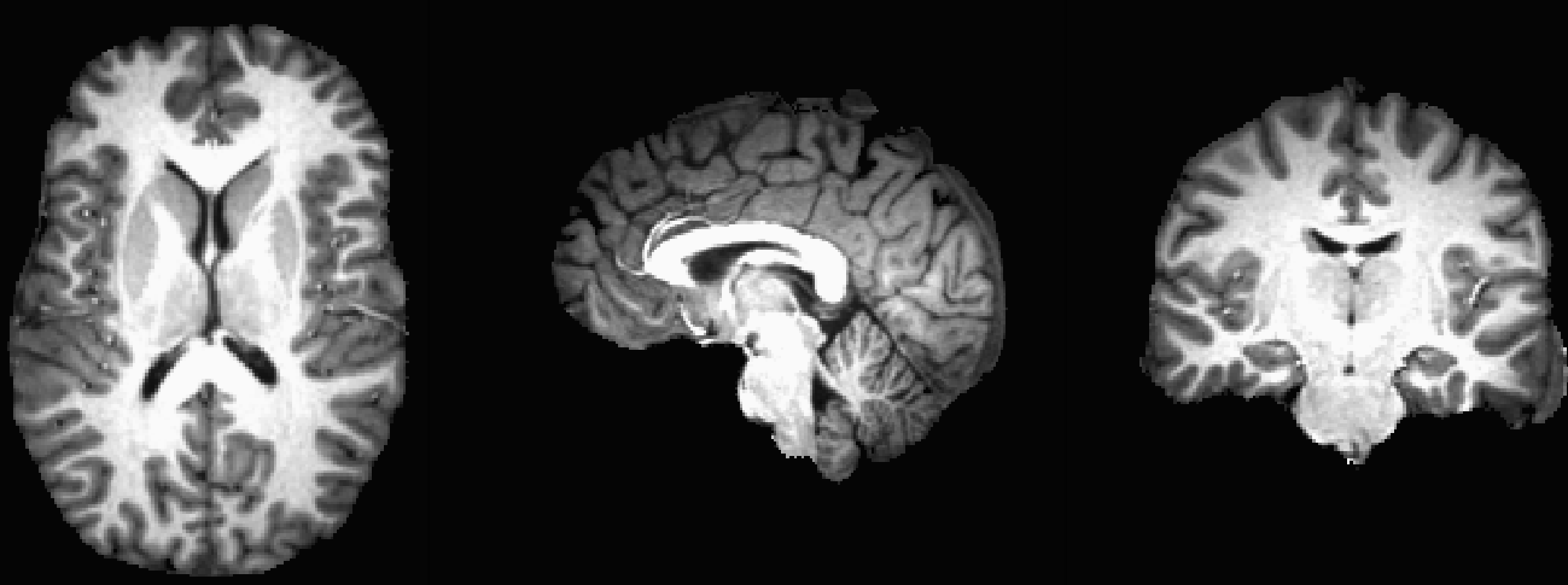
另一种查看条纹质量的方法是加载原始解剖图像sub-08_T1w.nii.gz作为Underlay,加载颅骨去除图像skull_strip_out作为Overlay。我们可以通过点击观察窗口中的任意位置,然后按 “o"键来交替查看和隐藏叠加;另一个选项是按 “u"键来在每张图像之间切换。这些查看选项将有助于您在预处理步骤前后检查数据。
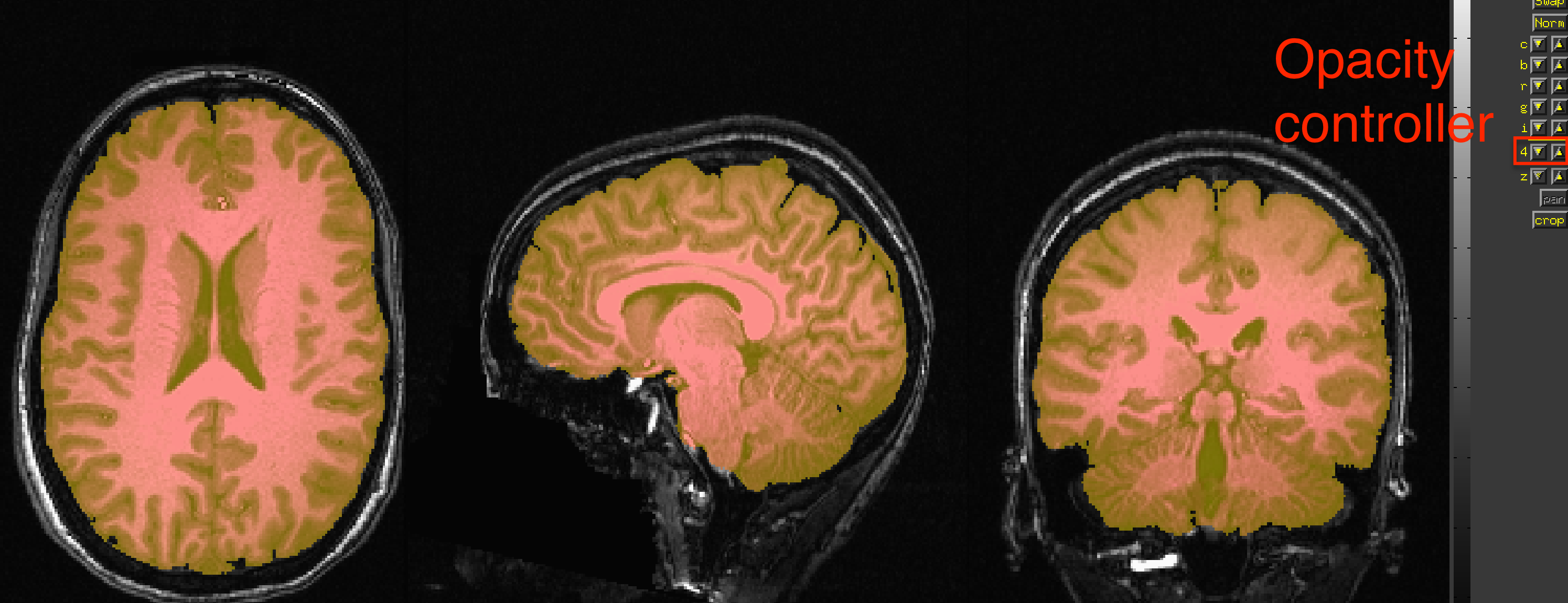
查看在原始解剖图像上叠加的头骨剥离图像的示例。图像的不透明度可通过右侧的控制器面板进行切换。
尽管颅骨剥离效果相当不错,而且对于大多数用途来说可能也没有问题,但让我们看看是否可以通过使用帮助文件中指定的任何选项来改进它。如果你仔细阅读帮助文件,你会注意到一个选项-push_to_edge,它可以帮助我们避免移除皮层的任何部分。一般来说,最好是将小块硬脑膜和其他非脑物质包括在内,而不是去除任何部分的大脑皮层。添加-prefix选项来标记输出结果也是非常有用的。键入以下命令:
3dSkullStrip -push_to_edge -input sub-08_T1w.nii.gz -prefix anat_ss
其中ss代表 “skull-stripped”。
几分钟后,将生成一个名为anat_ss+orig的新文件。在AFNI查看器中查看之前和之后的图像,并与之前生成的骷髅剥离图像进行比较。你发现有什么看起来更好吗?更差?你会选择哪一个头骨剥离图像,为什么?
uber_subjects.py脚本介绍
在接下来的章节中,我们将介绍典型的fMRI分析流水线的其他预处理步骤;不过,首先我们应该熟悉一个命令,它可以创建一个脚本来为运行所有这些预处理步骤:AFNI的uber_subject.py。
从技术上讲,uber_subject.py是一个图形用户界面的封装器,也就是说,它接收用户指定的所有图形用户界面输入,并将其导入另一个名为afni_proc.py的封装器。后一条命令生成一个大脚本,其中包含运行预处理每一步所需的每一条AFNI命令。
在命令行中输入uber_subject.py并按回车键。您将看到如下内容
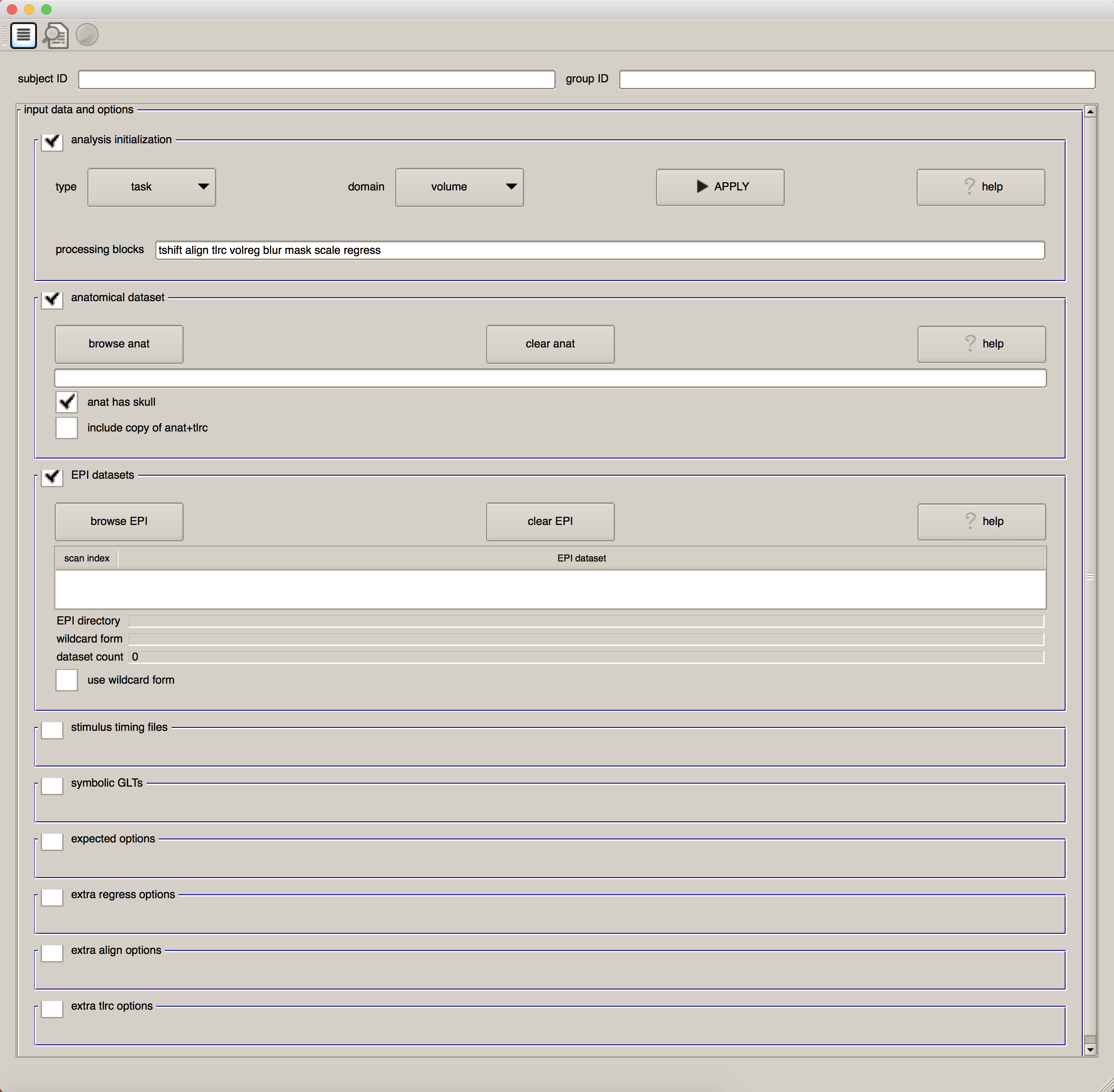
在接下来的章节中,我们将详细研究该图形用户界面的各个部分。现在,请注意第一个复选框 “Analysis Initialization”。默认情况下,数据分析类型为 “task”(而不是 “rest”,因为没有任务需要被试执行),域(domain)为"volume”(而不是 “surface”,我们将在后面关于相关程序SUMA的章节中讨论)。在 “preprocessing blocks"后面的字段:tshift、align、tlrc、volreg、blur、mask、scale 和 regress,我们可能看不懂。(如果这是我们第一次使用AFNI,我想它们看起来会很奇怪)。我们的任务是了解这些单词的含义、它们对应的预处理步骤以及为什么要这样做。
随着您对AFNI的熟练掌握,我们将能够更好地根据自己的需要改变分析的细节;但现在,我们以如何使用uber_subject.py创建一个预处理脚本为例。
练习
- 在
3dSkullStrip中使用-no_avoid_eyes或者-use_skull选项。查看帮助文件了解它们的作用,并预测它们将如何影响颅骨去除效果。你的预测是否与你在输出中看到的一致? - 将解剖图像载入AFNI查看器并按下 “Graph"按钮。这与您用功能图像查看 “Graph"输出有何不同?为什么?
运行Uber被试脚本:a. 配置分析脚本
现在我们可以使用uber_subject.py图形用户界面运行这些预处理步骤。现在,我们将简单地设置脚本并运行它;当它运行时,我们将阅读以下章节,这些章节描述了每个预处理步骤的作用。最后,我们将查看每个步骤的输出结果,并确定是否需要更改任何步骤。
首先,打开终端,导航到sub-08目录,输入uber_subject.py &。
逗号符号("&")将在后台执行您指定的命令;也就是说,它将在运行命令的同时保持终端可用,以便键入更多命令。如果执行的命令不带"&“符号,则可以通过单击执行命令的终端并键入ctrl+z,将当前正在运行的命令推至后台,但这将暂停当前命令;要将其置于后台(即继续运行该命令,同时保持终端空闲),请在终端中键入bg并按 Enter。
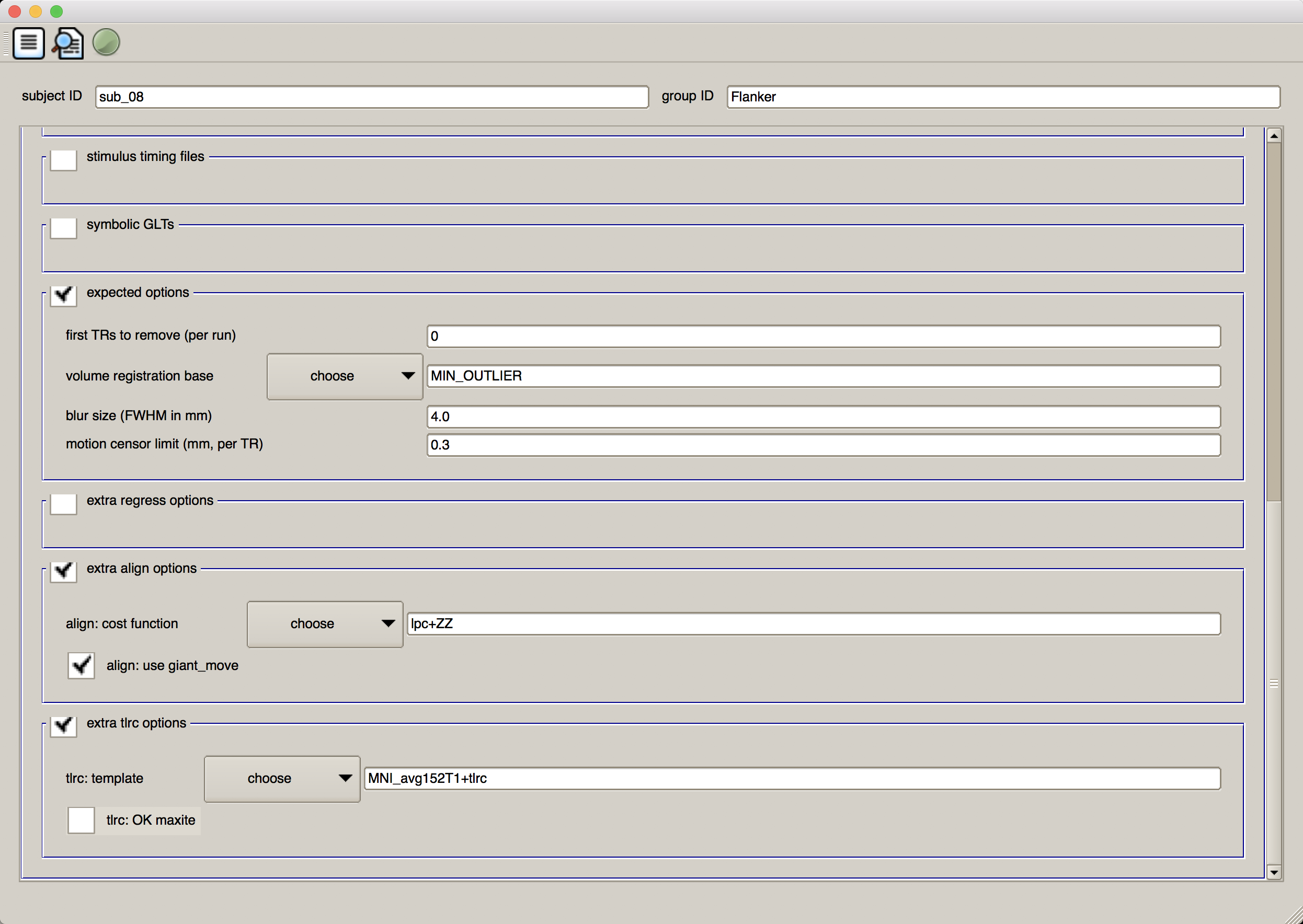
在"subject ID"字段中输入sub_08,在"group ID” 字段中输入Flanker。点击 “Analysis Initialization(分析初始化)“旁边的方框,删除"regress"块。(稍后当我们为每个受试者运行一般线性模型时,我们会将其包括在内)。然后点击 “anatomical dataset(解剖数据集)“部分的"browse anat"按钮,导航到anat目录,选择文件sub-08_T1w.nii.gz。点击"EPI datasets"部分的browse EPI按钮选择功能数据集,导航至func目录,按住shift并点击选择文件sub-08_task-flanker_run-1_bold.nii.gz和sub-08_task-flanker_run-2_bold.nii.gz。图形用户界面的前半部分应该如下所示:
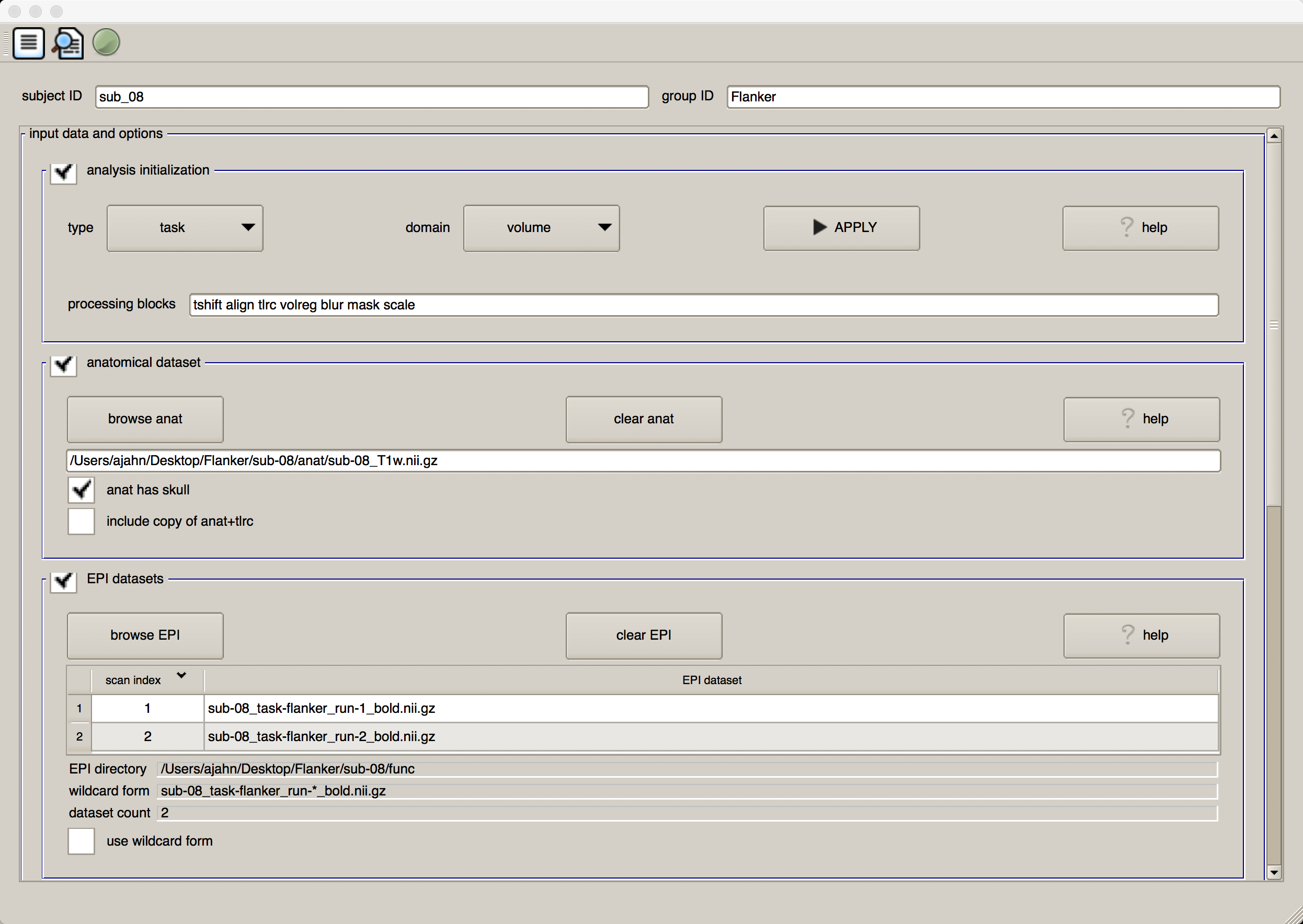
我们将跳过"stimulus timing files"和 “symbolic GLTs"部分,因为我们还没有进行回归。“expected options"字段中的默认值没有问题–不会删除任何TR,变异性最小的体块将用作配准的参考图像,4mm的平滑核将应用于数据,任何从TR到TR的合并移动量为0.3mm的体块将在审查文件(censor file)中标记(稍后将在回归过程中使用)。
运行Uber被试脚本:b. 运行分析脚本
当我们完成分析设置后,可以从左到右点击 GUI 窗口顶部的三个图标来执行分析。第一个图标看起来像一张纸,上面有线条;这将生成afni_proc.py命令,包含您在图形用户界面中指定的所有内容。点击图标,将返回两个窗口:一个窗口列出了每个从默认值修改的选项,并列出了每个输入,另一个窗口显示了afni_proc.py命令的代码。看看afni_proc.py命令中列出的命令和选项是如何与我们在uber_subject.py图形用户界面中输入的选项相对应的:
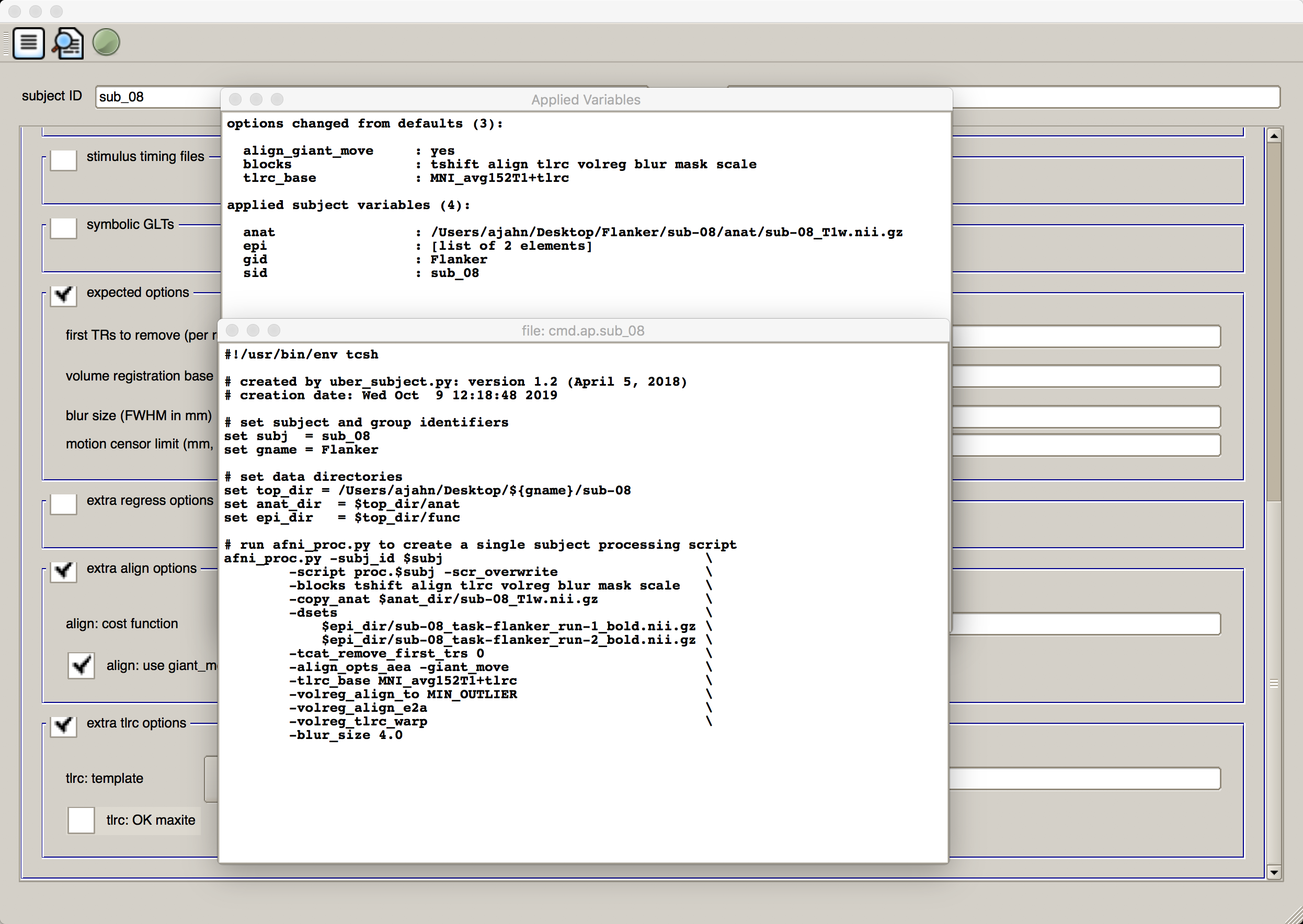
您将看到一条警告信息,上面写着 “** warning: no stim timing files given (resting state?)” 由于我们只是运行预处理,因此可以忽略该信息并点击 “OK(确定)"。
查看完输出窗口后,关闭它们。现在点击下一个图标,即一张纸上的放大镜。这将执行上一个窗口中列出的afni_proc.py代码,并返回我们应该注意的任何警告或错误(本案例子中暂无)。我们还将看到几行代码,指定如何运行该命令的输出,这是一个名为proc.sub_08的文件。
关闭窗口,回到终端。在sub-08目录中,注意到有一个新的目录结构已经创建。在下一个目录subject_results中,你会看到group.Flanker,下面是subj.sub_08。输入cd subject_results/group.Flanker/subj.sub_08,导航到该目录。该目录下有三个文件:
cmd.ap.sub_08:这是由uber_subject.py生成的afni_proc.py文件。output.cmd.ap.sub_08:这是afni_proc.py的输出,其中会生成我们应该注意的警告或错误。proc.sub_08:将执行实际预处理的原始AFNI命令,由afni_proc.py命令编译。用vi或其他文本工具打开该文件,检查其中的内容。它应该是这样的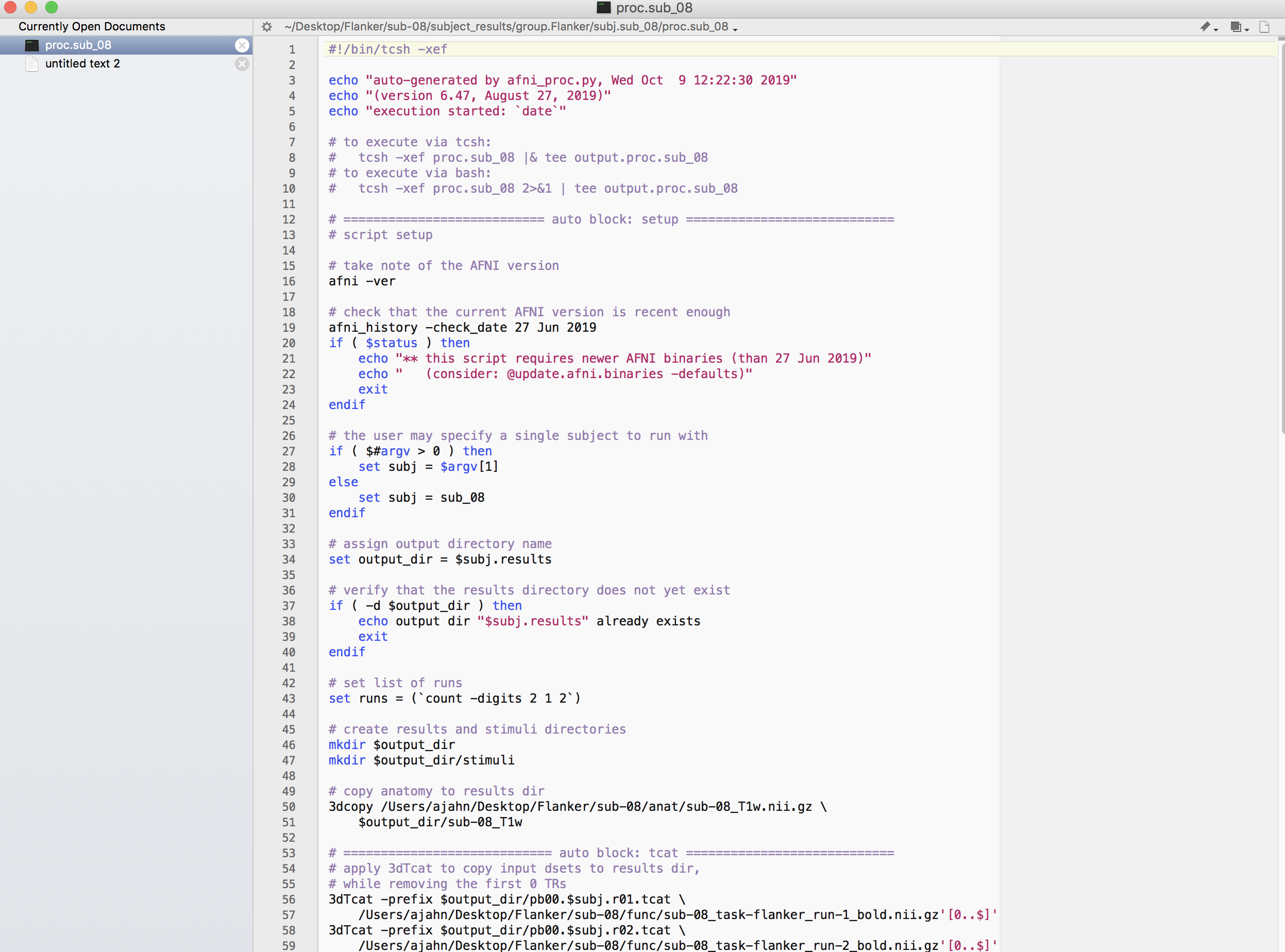
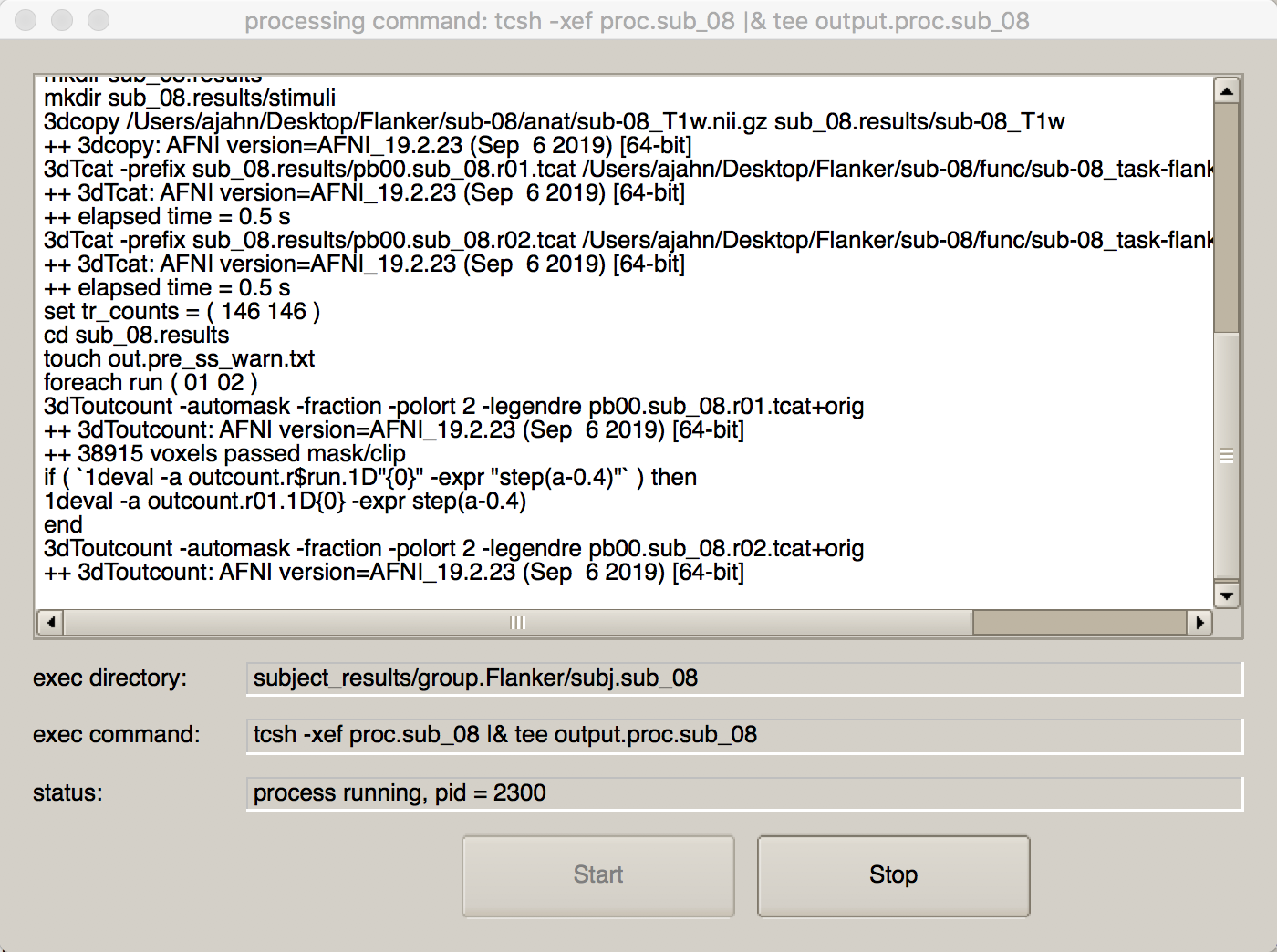
现在我们可以运行该脚本了。我们可以键入output.cmd.ap.sub_08中列出的代码,或者直接按uber_subject.py GUI 顶部的绿色图标按钮。如果你按了后者,就会看到另一个窗口打开,显示正在运行的每条命令及其相应的输出:
如果uber_subject.py不能使用,比如pyqt4老旧安装麻烦等问题,导致脚本不能使用,建议使用下述使用uber_subject.py脚本输出的sub_08_afni_proc.sh脚本。
#!/usr/bin/env tcsh
# created by uber_subject.py: version 1.2 (April 5, 2018)
# creation date: Mon Nov 18 12:30:05 2019
# set subject and group identifiers
set subj = sub_08
set gname = Flanker
# set data directories
set top_dir = ${PWD}/sub-08
set anat_dir = $top_dir/anat
set epi_dir = $top_dir/func
set stim_dir = $top_dir/func
# run afni_proc.py to create a single subject processing script
afni_proc.py -subj_id $subj \
-script proc.$subj -scr_overwrite \
-blocks tshift align tlrc volreg blur mask scale regress \
-copy_anat $anat_dir/sub-08_T1w.nii.gz \
-dsets \
$epi_dir/sub-08_task-flanker_run-1_bold.nii.gz \
$epi_dir/sub-08_task-flanker_run-2_bold.nii.gz \
-tcat_remove_first_trs 0 \
-align_opts_aea -giant_move \
-tlrc_base MNI_avg152T1+tlrc \
-volreg_align_to MIN_OUTLIER \
-volreg_align_e2a \
-volreg_tlrc_warp \
-blur_size 4.0 \
-regress_stim_times \
$stim_dir/congruent.1D \
$stim_dir/incongruent.1D \
-regress_stim_labels \
congruent incongruent \
-regress_basis 'GAM' \
-regress_censor_motion 0.3 \
-regress_motion_per_run \
-regress_opts_3dD \
-jobs 8 \
-gltsym 'SYM: incongruent -congruent' -glt_label 1 \
incongruent-congruent \
-gltsym 'SYM: congruent -incongruent' -glt_label 2 \
congruent-incongruent \
-regress_reml_exec \
-regress_make_ideal_sum sum_ideal.1D \
-regress_est_blur_epits \
-regress_est_blur_errts \
-regress_run_clustsim no
2. 时间层校正(Slice-Timing Correction)
整个照片是在一瞬间拍摄完成的,而fMRI的volume是分片(slices)获取的。每个切片都需要时间来获取(从几十到几百毫秒)。
两种最常用的创建volume的方法是顺序和交错的切片采集。顺序切片采集是连续采集每个相邻的切片,从下到上或从上到下。交错式切片采集每一个间隔的切片,然后在第二遍的时候填补空隙。这两种方法在下面的视频中都有说明。

正如在后面看到的,当我们对每个体素的数据进行建模时,我们假设所有的切片(slices)是同时获得的。为了使这一假设有效,每个切片的时间序列需要在时间上向后移动,即获取该切片所需的时间。Sladky等人(2011年)还证明,对于TR较长(如2s或更长)的研究,尤其是大脑背侧区域的研究,切片时间校正可使统计能力显著提高。
尽管时间层校正似乎是合理的,但也有一些反对意见:
- 一般来说,除非需要,否则最好不要对数据进行插值(即编辑);
- 对于短的TR(例如,大约1秒或更短),时间层校正似乎不会导致统计能力的任何明显提高;
- 许多由时间层校正解决的问题可以通过在统计模型中使用时间导数(temporal direivate)来解决(在后面关于模型拟合的章节中讨论)。
现在,我们将以第一个切片为基准进行切片定时校正(由3dTshift命令的-tzero 0选项指定)。(运行时间层校正的代码可在proc脚本的第 97-100行找到:
foreach run ( $runs )
3dTshift -tzero 0 -quintic -prefix pb01.$subj.r$run.tshift \
pb00.$subj.r$run.tcat+orig
end
这将以第一个切片为参照,对每次run进行时间层校正。(请记住,在AFNI中,所有内容的索引都是从0开始的,也就是说,在这种情况下,0代表volume的第一个切片)。该命令还使用了一个名为-quintic的选项,它使用五次多项式对每个切片进行重新采样。换句话说,由于我们需要替换一个切片内的体素值,因此可以通过使用更多其他切片的信息来提高精确度。这确实在一定程度上引入了切片之间的相关性,我们稍后将尝试使用3dREMLfit对数据进行预白化(即去相关性)来纠正这种相关性。
3. 配准与归一化(Registration and Normalization)
虽然大多数人的大脑是相似的–例如,每个人都有扣带回(cingulate gyrus)和胼胝体(corpus callosum)–但在大脑的大小和形状上也有差异。因此,如果我们想做一个群体分析(group analysis),我们需要确保每个受试者的每个体素都对应于大脑的同一部位。例如,如果我们要测量视觉皮层中的一个体素,我们要确保每个受试者的视觉皮层都是一致的。
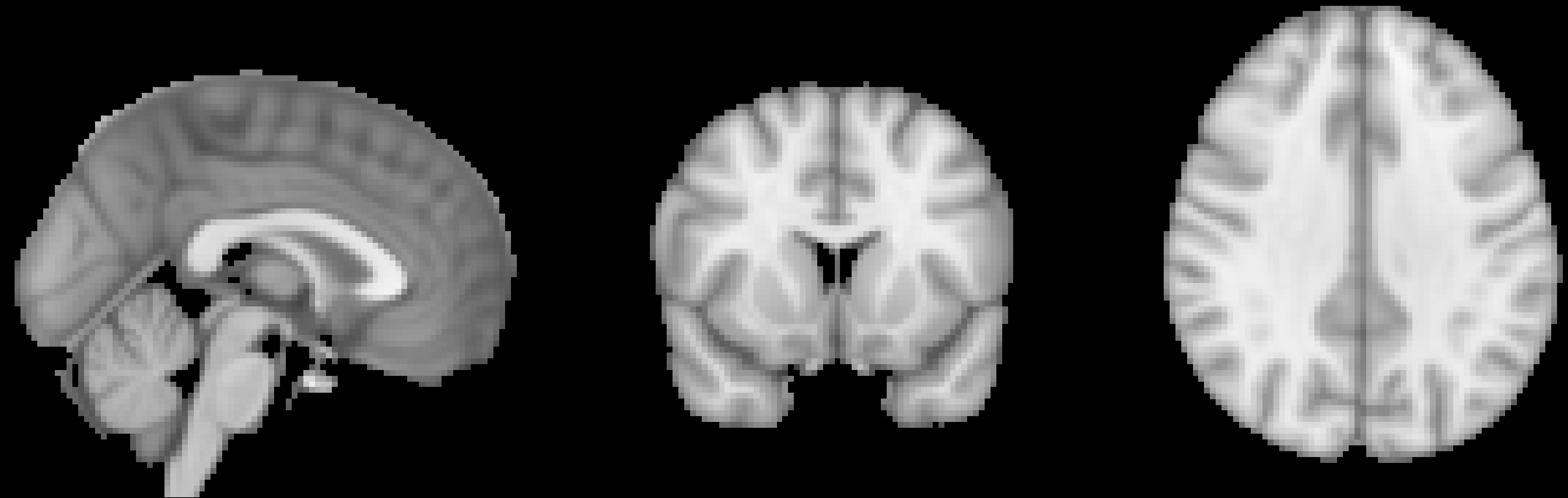
这是通过对图像进行配准(Registering)和归一化(Normalizing)来实现的。就像你要折叠衣服以装入行李箱一样,每个大脑都需要被转换为具有相同的大小、形状和尺寸。我们通过归一化(或扭曲)到模板来实现这一目标。模板是一个具有标准尺寸和标准坐标的大脑,因为大多数研究人员都同意在报告他们的结果时使用它们。这样一来,如果你将你的数据归一到该模板,并在坐标X=3、Y=20、Z=42处发现了一个效应,那么其他将他们的数据扭曲到同一模板的人可以将他们的结果与你的结果进行对照。模板大脑的尺寸和坐标也被称为标准化空间(standardized space)。
仿射变换(Affine Transformations)
为了将图像扭曲成一个模板,我们将使用一个仿射变换(Affine transformation)。这类似于运动校正中描述的刚体变换,但它又增加了两个变换:缩放(zooms)和错切(shear)。平移和旋转对于像笔这样的日常物体来说是很容易做到的,而缩放和错切则更不寻常–缩放可以缩小或放大图像,而错切则是将图像的对角线相对的角拉开。下面的动画总结了这四种类型的线性变换。

与刚体变换一样,缩放和错切都有三个自由度: 你可以沿x轴、y轴或z轴缩放或错切图像。那么,总的来说,仿生变换有12个自由度((平移、旋转、缩放、错切)* (x、y、z)= 4 * 3= 12)。这些也被称为线性变换,因为沿轴的一个方向应用的变换会伴随着相反方向的等量变换。例如,向左平移一毫米,意味着图像从右边移动了一毫米。同样地,如果一个图像沿Z轴放大一毫米,它就会沿该轴的两个方向放大一毫米。没有这些约束的变换被称为非线性变换。例如,非线性变换可以在一个方向上放大图像,而在另一个方向上缩小图像,就像挤压海绵时一样。这些类型的变换将在后面讨论。
配准与归一化(Registration and Normalization) 回顾一下,我们的数据集中既有解剖学图像,也有fMRI图像。我们的目标是将fMRI图像变换到模板上,这样我们就可以对所有的受试者进行组分析。虽然简单地将fMRI图像直接翘曲到模板上似乎是合理的,但在实践中,这样做效果并不好–图像是低分辨率的,因此不太可能与模板的解剖细节相匹配。解剖图像是一个更好的选择。
虽然这似乎对我们实现目标没有帮助,但事实上,对解剖图像进行扭曲可以帮助将fMRI图像带入标准化的空间。请记住,解剖和功能扫描通常是在同一时段获得的,而且在两次扫描之间,受试者的头部几乎没有移动。如果我们已经将解剖图像规范化为一个模板,并记录了所做的转换,我们就可以将同样的转换应用于fMRI图像–只要它们与解剖图像在同一位置开始。
这种功能图像和解剖图像之间的对位被称为配准。大多数配准算法使用以下步骤:
- 假设功能图像和解剖图像处于大致相同的位置。如果它们不在同一位置,则对准图像的轮廓。
- 利用解剖图像和功能图像具有不同的对比度权重这一事实–也就是说,在解剖图像上图像较暗的区域(如脑脊液)在功能图像上会显得明亮,反之亦然。这被称为互信息(mutual information)。配准算法移动图像以测试解剖和功能图像的不同重叠,将一个图像上的明亮体素与另一个图像上的黑暗体素相匹配,将黑暗体素与明亮体素相匹配,直到找到一个无法改进的匹配。
- 一旦找到了最佳匹配,然后将用于将解剖图像与模板进行扭曲的相同转换应用于功能图像。

通过AFNI的align_epi_anat.py来配准
align_epi_anat.py命令可以同时完成多个预处理步骤–配准、将功能图像的体积对齐和时间层校正。但在本例中,我们将只用它进行配准。这一步骤的代码可以在 proc 脚本的第 110-115 行找到:
align_epi_anat.py -anat2epi -anat sub-08_T1w+orig \
-save_skullstrip -suffix _al_junk \
-epi vr_base_min_outlier+orig -epi_base 0 \
-epi_strip 3dAutomask \
-giant_move \
-volreg off -tshift off
align_epi_anat.py -anat2epi -anat anat/sub-08_T1w.nii.gz \
-save_skullstrip -suffix _al_junk \
-epi func/sub-08_task-flanker_run-1_bold.nii.gz -epi_base 0 \
-epi_strip 3dAutomask \
-giant_move \
-volreg off -tshift off
第一个选项-anat2epi表示解剖图像将与功能图像对齐,反之亦然。一般来说,我们希望尽可能减少对功能数据的改动和插值。因此,如果需要对图像进行移动和轻微变形,我们会选择在解剖图像上进行。
-suffix命令将_al_junk字符串附加到一些配准的中间阶段,我们稍后将用它来对功能图像进行归一化处理。epi选项(即-epi、-epi_base 和-epi_strip)表示将使用变化最小的功能容积(functional images)作为参考图像,并使用3dAutomask(3dSkullStrip的替代方法)去除非脑组织。-giant_move试图在解剖图像和功能图像之间找到良好的初始配准;最后两个选项表示我们不想在当前命令中包含配准和时间层校正。
通过AFNI的@auto_tlrc来归一化
对齐解剖和功能图像后,我们首先要将解剖图像归一化为模板。在下一章中将看到,这些扭曲也将应用于功能图像。要对解剖图像进行归一化处理,我们将使用@auto_tlrc命令;该命令和下面的cat_matvec命令位于 proc 脚本的第 118-122 行:
# warp anatomy to standard space: 将解剖图转到标准空间
$ @auto_tlrc -base MNI_avg152T1+tlrc -input anat/sub-08_T1w_brain.nii.gz -
no_ss
Copying NIFTI volume to anat/sub-08_T1w_brain_AFN_OYmeqfZ3JqF7n9LNC7gDAA
++ 3dcopy: AFNI version=AFNI_23.1.10 (Jun 29 2023) [64-bit]
Error: input dataset must be in the current directory
Current path for input dataset is anat
Sorry.
++ anat already stripped, re-using
*********** Warning *************
Dataset centers are 92.736319 mm
apart. If registration fails, or if
parts of the original anatomy gets
cropped, try adding option
-init_xform AUTO_CENTER
to your @auto_tlrc command.
*********************************
Padding ...
++ 3dZeropad: AFNI version=AFNI_23.1.10 (Jun 29 2023) [64-bit]
++ output dataset: ./__ats_tmp__ref_MNI_avg152T1_15pad+tlrc.BRIK
...
++ Authored by: RW Cox
++ Processing AFNI dataset __ats_tmp___upad15_sub-08_T1w_brain_AFN_OYmeqfZ3JqF7n9LNC7gDAA+tlrc
+ setting Warp parent
++ 3drefit processed 1 datasets
++ 3dAFNItoNIFTI: AFNI version=AFNI_23.1.10 (Jun 29 2023) [64-bit]
Cleanup ...
# store forward transformation matrix in a text file:将正向变换矩阵存储到文本文件中
$ cat_matvec sub-08_T1w_ns+tlrc::WARP_DATA -I > warp.anat.Xat.1D
第一条命令表示以图像MNI_avg152T1为模板,以头骨剥离的解剖图像为源图像,或移动图像使其与基础图像或参考图像最匹配。-no_ss选项表示解剖图像已经过头骨切片处理。
为了使模板和解剖图像对齐,解剖图像需要通过上述变换进行移动和转换。这将产生一系列数字,组成仿射变换矩阵(affine transformation matrix),并存储在解剖图像的标题中。第二条命令cat_matvec将提取该矩阵并复制到一个名为warp.anat.Xat.1D的文件中。下一节将介绍如何使用该矩阵将功能图像转换到相同的归一化空间。
练习
- 在上述
@auto_tlrc命令中,默认模板是MNI_avg152+tlrc,可以在~/abin目录中找到。输入ls ~/abin | grep MNI,查看 MNI 空间中的可用模板。并不是所有模板都能用于 T1 解剖扫描的归一化,但其中有几个模板是可以的,包括MNI152_1mm_uni+tlrc和MNI152_T1_2009c+tlrc。对解剖图像进行颅骨切片后,请尝试使用以下命令:
$ 3dcopy sub-01_T1w_ns+orig. T1w_cop @auto_tlrc -base MNI152_T1_2009c+tlrc -input T1w_copy+orig -no_ss
观察输出结果与上述原始命令有何不同。也用MNI152_T1_2009c+tlrc模板试试,在 orig 空间创建原始 T1 加权解剖图像的新副本。你更喜欢使用哪种模板?为什么?另外,为什么你认为默认是MNI_avg152T1+tlrc大脑? 提示:在 AFNI 查看器中输入afni ~/abin查看每个模板。
4. 对齐与运动校正(Alignment and Motion Correction)
简介
如果我们尝试过给移动的物体拍照,通常图像会很模糊。相反,如果拍摄时物体保持静止不动,那么就能获得更加清晰、轮廓分明的图像。
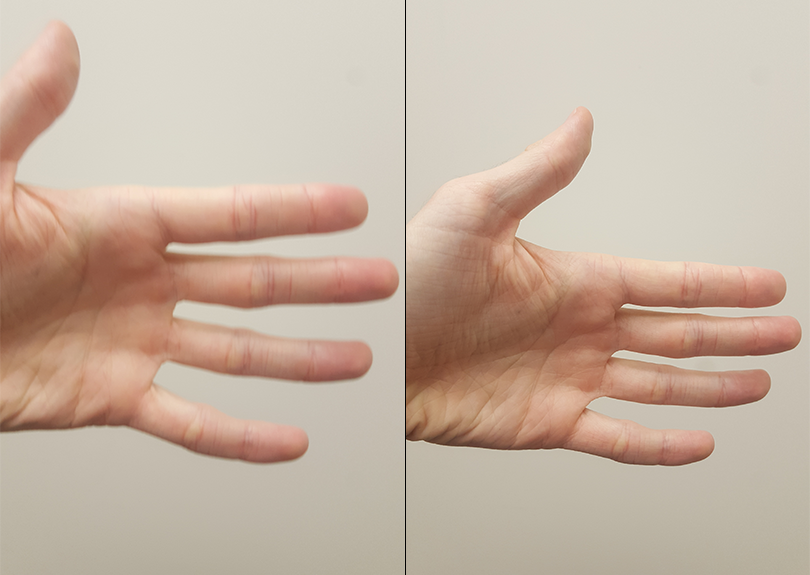
当我们拍摄大脑的三维图像时,概念也是一样的。如果拍摄对象在移动,图像看起来就会模糊;如果拍摄对象静止不动,图像看起来就不会那么模糊,轮廓会更加清晰。但这还不是全部:如果实验对象移动频繁,我们也有可能测量到移动体素的信号。这样,我们就有可能在部分实验中测量到来自该体素的信号,而在实验对象移动后,又测量到来自不同区域或组织类型的信号。
由于运动会产生信号,这些运动也会给成像数据带来混淆。如果受试者每次都对刺激做出反应而移动–例如,受试者每次感受到电击时都会扭动头部–那么就无法确定我们测量的信号是对刺激做出的反应,还是因为移动而产生的。
刚体变换(Rigid-Body Transformations) “撤销"这些运动的方法之一是进行刚体变换。为了说明这一点,请拿起附近的一个物体:例如手机或咖啡杯。把它放在你面前,并在脑海中标出它的位置。这就是参考点。然后将物体向左移动一英寸。这就是所谓的平移(translation),也就是向左或向右、向前或向后、向上或向下的任何移动。如果想让物体回到起点,只需向右移动一英寸即可。
同样,如果你将物体向左或向右旋转,也可以通过向相反方向等量旋转来撤销。这些被称为旋转(rotation),和平移一样,它们有三个自由度,或者说有三种移动方式:绕 x 轴(也称为俯仰,或前后倾斜)、绕 y 轴(也称为滚动,或左右倾斜)和绕 z 轴(或偏航,如摇头说 “不”)。
通过移动自己的头部来尝试这些平移和旋转。首先,直视前方时左右移动头部(沿 x 轴平移)。然后,前后(y 轴)上下(z 轴)移动头部。
我们对volumes进行同样的操作。我们将时间序列中的第一个volume称为reference volume(参考容积),而不是上面例子中使用的参考点。如果在扫描过程中,被扫描者的头部向右移动了一英寸,我们就可以检测到这一移动,并通过将volume向左移动一英寸来消除这一移动。这样做的目的是检测到任何一个容积的移动,并将这些容积与参考容积重新对齐。

AFNI的3dvolreg
AFNI 中的运动校正是通过3dvolreg命令完成的。在由uber_subject.py生成的典型分析脚本中,会有一个代码块,其标题为 ======volreg======。该代码块中有几个命令,但对我们现在的目的来说,最重要的是以 “3dvolreg"开头的一行。
你会看到该命令使用了几个选项。-base选项指的是参考体;在本例中,参考图像是由之前的3dToutcount命令确定的体素中异常值最少的体。-1Dfile 命令会将运动参数写入一个文本文件,并在文件后附加"1D”,而-1Dmatrix_save则会保存一个仿射矩阵,该矩阵会显示为了与参考图像相匹配,每个 TR 在每个仿射维度上需要 “warped"的程度。
# register and warp
foreach run ( $runs )
# 将每个volume配准到基准图像
3dvolreg -verbose -zpad 1 -base vr_base_min_outlier+orig \
-1Dfile dfile.r$run.1D -prefix rm.epi.volreg.r$run \
-cubic \
-1Dmatrix_save mat.r$run.vr.aff12.1D \
pb01.$subj.r$run.tshift+orig
然后,将该仿射变换矩阵与在将解剖数据集扭曲到归一化空间期间以及将解剖数据集与fMRI数据配准时生成的仿射变换矩阵连接起来:
# catenate volreg/epi2anat/tlrc xforms
cat_matvec -ONELINE \
{$subj}_T1w_ns+tlrc::WARP_DATA -I \
{$subj}_T1w_al_junk_mat.aff12.1D -I \
mat.r$run.vr.aff12.1D > mat.r$run.warp.aff12.1D
此连接仿射矩阵与3dAllineate命令一起使用,可在一个步骤中创建运动校正和归一化的 fMRI 数据集(使用1Dmatrix_apply选项):
# 对 all-1数据集进行扭曲处理,以进行外延遮蔽(extents masking)
3dAllineate -base {$subj}_T1w_ns+tlrc \
-input rm.epi.all1+orig (输入 rm.epi.all1+orig
-1Dmatrix_apply mat.r$run.warp.aff12.1D \
-mast_dxyz 3 -final NN -quiet\
-prefix rm.epi.1.r$run
这些代码块一开始可能很难理解,但请始终牢记 AFNI 命令的基本结构: 命令名称后跟选项,通常还包括一个”-prefix"选项,用于标注输出结果。运动校正和归一化命令通常还包括一对”-base"和”-input"选项,用于标明哪个数据集正在与哪个参考数据集对齐。我们很可能不会编辑uber_subject.py生成的文件中的这些代码行,但了解它们的编写方式仍然很有用;如果我们愿意,可以在本脚本之外使用这些命令对齐其他数据集。
练习
- 在
3dvolreg命令中,将-base改为 0,观察输出结果与使用vr_base_min_outlier作为参考体积有何不同。再试一次,使用3dTstat创建该run的平均函数图像,并使用该平均函数图像作为基准;这与SPM12软件包中的做法类似。您更喜欢哪种方法,为什么?
5. 平滑(Smoothing)
常见的做法是对fMRI数据进行平滑处理,或将每个体素的信号替换为该体素邻近区域的加权平均。这初看起来很奇怪–为什么我们要使图像比原来更模糊?
的确,平滑化会降低fMRI数据的空间分辨率,而我们并不希望分辨率降低。但平滑化也有好处,这些好处可以超过缺点。例如,我们知道fMRI数据包含很多噪音,而且噪音经常大于信号。通过对附近的体素进行平均化,我们可以消除噪音并增强信号。

平滑处理是通过 AFNI 的 3dmerge 命令完成的,您可以在 proc_Flanker 脚本的 “blur “标题下找到该命令(第 216-221 行)。在所有预处理步骤中,这个步骤使用的代码行数最少:
# 模糊每个run的每个volume
foreach run ( $runs )
3dmerge -1blur_fwhm 4.0 -doall -prefix pb03.$subj.r$run.blur \
pb02.$subj.r$run.volreg+tlrc
end
-1blur_fwhm选项指定了平滑图像的量,单位为毫米–在本例中为4毫米。-doall选项会将平滑核应用到图像中的每个卷,而-prefix选项会一如既往地指定输出数据集的名称。
最后的预处理步骤是将这些平滑图像缩放为平均信号强度为100的图像,这样就可以用信号变化百分比来衡量与平均值的偏差。然后用掩膜去除任何非脑体素,这些图像就可以进行统计分析了。要了解 AFNI 如何完成最后两个预处理步骤,请查看第6小节掩膜与缩放。
练习
- 导航到
sub-01/func目录,输入3dmerge -1blur_fwhm 4.0 -doall -prefix test_blur_4mm.nii sub-01_task-flanker_run-1_bold.nii.gz对原始功能数据应用不同的平滑核。在 AFNI GUI 中查看输出文件。然后,用 8mm、12mm 和 20mm 的平滑内核做同样的操作,记住每次平滑时都要更改输出文件的名称。完成后,输入rm test*即可删除所有平滑图像。
6. 掩膜(or 掩码)与缩放(Masking and Scaling)
a.什么是掩膜(掩码,or masking)? 正如在之前的教程中所看到的,一卷fMRI数据既包括大脑,也包括周围的头骨和颈部–我们对使用AFNI分析这些区域并不感兴趣,尽管它们与大脑体素一样,包含有时间序列数据的体素。此外,尽管乍一看可能并不明显,但我们有大量的体素包含在头部以外的空气中。
为了减少数据集的大小,从而加快分析速度,我们可以对数据应用掩码。掩码简单地表示哪些体素要进行分析–掩码内的体素保留其原始值(或赋值为 1),而掩码外的体素赋值为 0。这就好比用描图纸描出一幅画的轮廓,然后沿线剪切,保留线内的部分,舍弃其余部分。应用到fMRI数据中,掩码之外的任何东西都会被我们认为是噪音或不感兴趣的东西。
掩码使用AFNI的3dAutomask命令创建,它只需要输入和输出数据集的参数(proc_Flanker 脚本第 223-260 行):
foreach run ( $runs )
3dAutomask -prefix rm.mask_r$run pb03.$subj.r$run.blur+tlrc
end
掩码块中的其余代码将创建一个掩码集合,代表实验中所有单个fMRI数据集的范围。然后为解剖数据集计算一个掩码,再取fMRI掩码和解剖掩码的交集:
# create union of inputs, output type is byte
3dmask_tool -inputs rm.mask_r*+tlrc.HEAD -union -prefix full_mask.$subj
# ---- create subject anatomy mask, mask_anat.$subj+tlrc ----
# (resampled from tlrc anat)
3dresample -master full_mask.$subj+tlrc -input {$subj}_T1w_ns+tlrc \
-prefix rm.resam.anat
# convert to binary anat mask; fill gaps and holes
3dmask_tool -dilate_input 5 -5 -fill_holes -input rm.resam.anat+tlrc \
-prefix mask_anat.$subj
# compute tighter EPI mask by intersecting with anat mask
3dmask_tool -input full_mask.$subj+tlrc mask_anat.$subj+tlrc \
-inter -prefix mask_epi_anat.$subj
# compute overlaps between anat and EPI masks
3dABoverlap -no_automask full_mask.$subj+tlrc mask_anat.$subj+tlrc \
|& tee out.mask_ae_overlap.txt
# note Dice coefficient of masks, as well
3ddot -dodice full_mask.$subj+tlrc mask_anat.$subj+tlrc \
|& tee out.mask_ae_dice.txt
# ---- create group anatomy mask, mask_group+tlrc ----
# (resampled from tlrc base anat, MNI_avg152T1+tlrc)
3dresample -master full_mask.$subj+tlrc -prefix ./rm.resam.group \
-input /Users/ajahn/abin/MNI_avg152T1+tlrc
# convert to binary group mask; fill gaps and holes
3dmask_tool -dilate_input 5 -5 -fill_holes -input rm.resam.group+tlrc \
-prefix mask_group
我们将在下一节更详细地了解这段代码的输出结果,即创建一个掩码,用于追踪图像检测到的信号的轮廓:

从上图可以看出,掩码似乎排除了眶额皮层的某些部分。由于这些区域的信号一开始就相对较低(这是由于一种被称为磁感应伪影(magnetic susceptibility artifact)的现象导致信号丢失),掩码假定该区域不包含任何脑体素。这可以通过使用一种叫做场图反扭曲(field map unwarping)的技术来解决。这超出了本教程的范围,但如果您有兴趣,可以在此处了解一种此类方法。
b.缩放(scaling) fMRI数据的一个问题是,我们收集的数据单位是任意的,本身没有意义。我们收集到的信号强度会因运行的不同而不同,也会因受试者的不同而不同。要在受试者内部或受试者之间进行有用的比较,唯一的方法就是使用贝塔权重(beta weight)(将在稍后的统计一章中讨论)来表示不同条件下的信号强度对比。
为了使不同研究之间的信号强度对比也有意义,AFNI将每个体素的时间序列单独缩放至平均值100:
# scale each voxel time series to have a mean of 100
# (be sure no negatives creep in)
# (subject to a range of [0,200])
foreach run ( $runs )
3dTstat -prefix rm.mean_r$run pb03.$subj.r$run.blur+tlrc
3dcalc -a pb03.$subj.r$run.blur+tlrc -b rm.mean_r$run+tlrc \
-c mask_epi_extents+tlrc \
-expr 'c * min(200, a/b*100)*step(a)*step(b)' \
-prefix pb04.$subj.r$run.scale
end
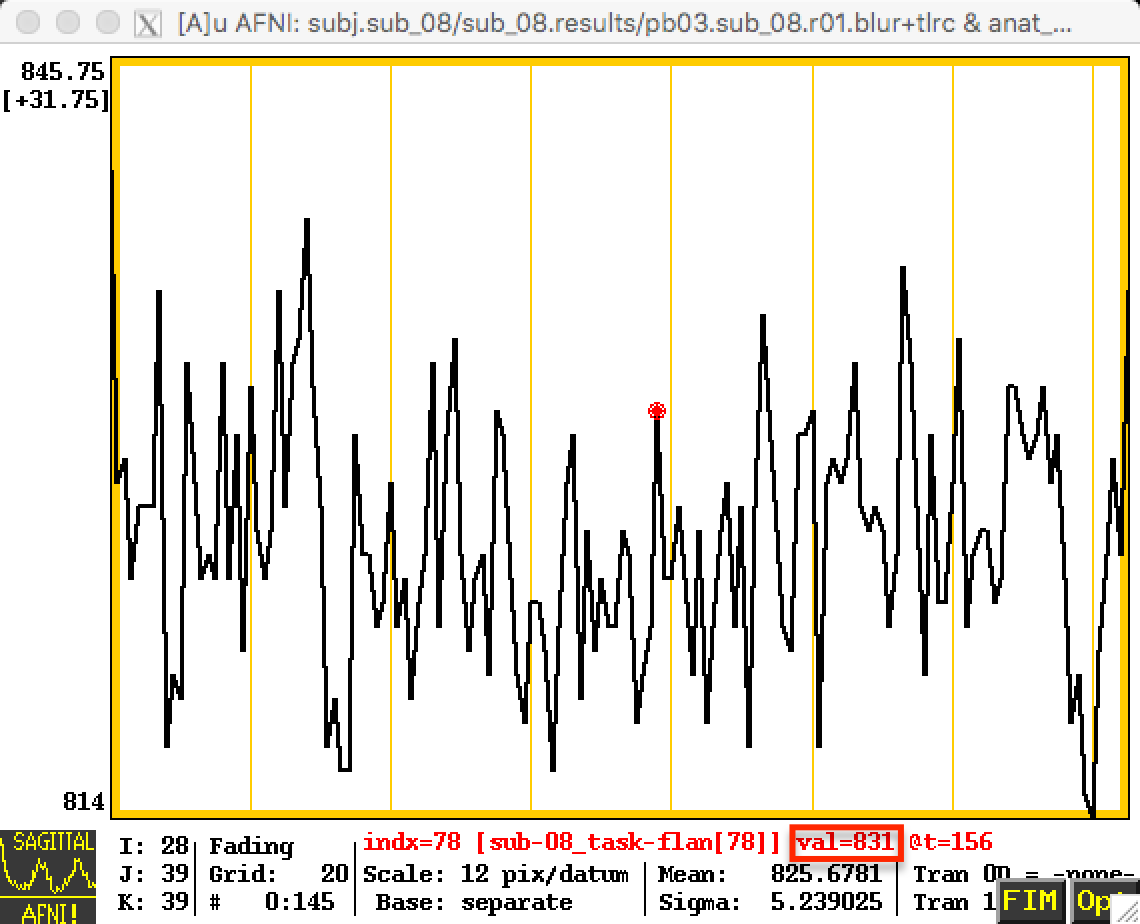
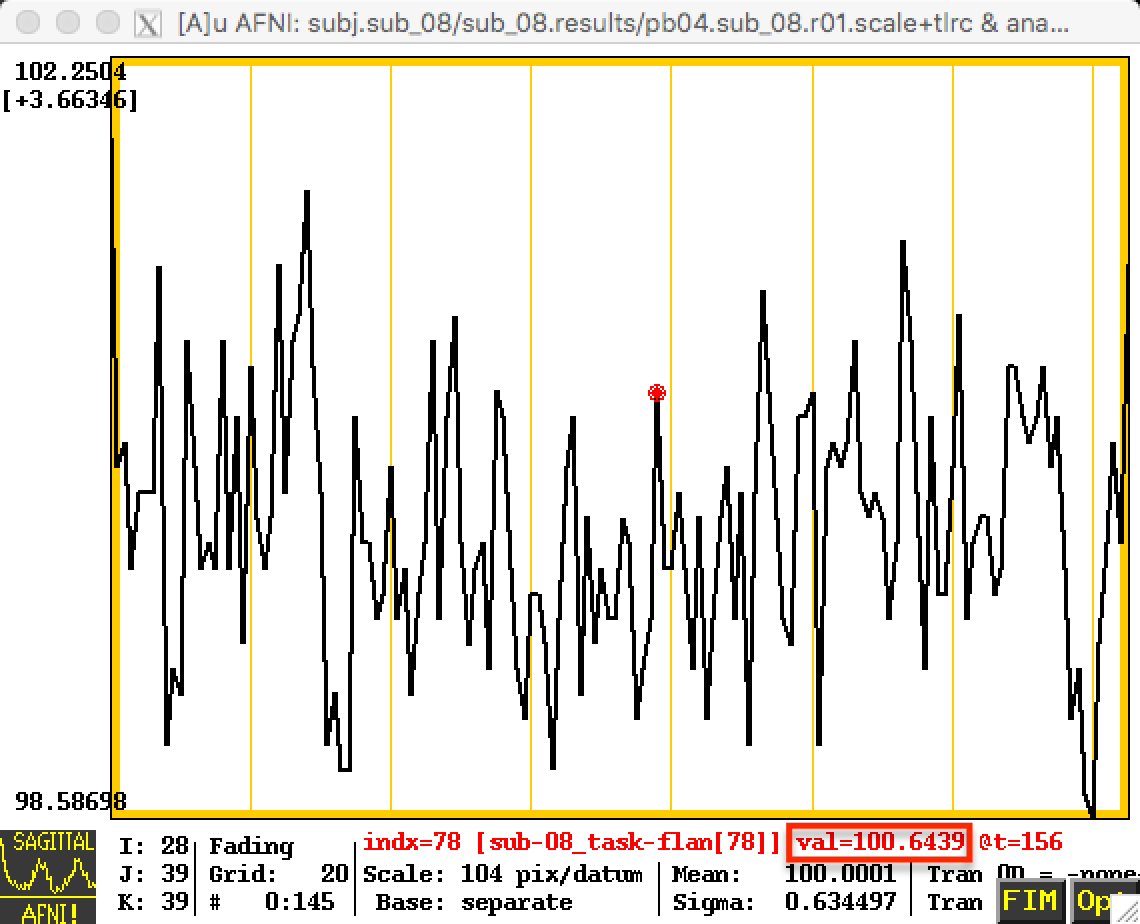
这些变化将反映在时间序列中;下面第一张图片是缩放前的时间序列,下一张图片显示的是缩放后的时间序列。请注意,第一张图片中的数值相对较高–在 800左右–而且这些数值是任意的;在另一个实验对象中,这些数值很可能在500或900左右。通过将每个受试者的数据缩放至相同的平均值(如第二张图片所示),我们可以将每个受试者的每次数据运行置于相同的刻度上。
现在我们已经完成了预处理步骤,是时候回顾每个步骤并检查数据质量了。在下一节中,我们将介绍如何完成这项工作。
7. 检查预处理结果
1).导航到已处理的数据目录
uber_subject.py生成的脚本完成后,导航到包含预处理数据的目录。默认情况下,AFNI将按照以下格式创建一个新的目录树:
subject_results/group.<GroupName>/subj.<subjName>/<subjName>.results
其中,GroupName和subjName指定在uber_subject.py GUI 的subject ID 和group ID 字段中。在这种情况下,我们可以通过键入以下内容导航到结果目录
$ cd subject_results/group.Flanker/subj.sub_08/sub_08.results
稍后,当我们讨论分析脚本或对数据集中的所有被试进行自动分析时,我们将通过修剪不必要的子目录来简化目录树。
该目录包含经过每一步预处理后的不同图像版本。例如,包含字符串pb01(即Processing Block 01)和字符串 tshift 的文件表示这些图像已使用3dTshift命令进行了切片时间校正。
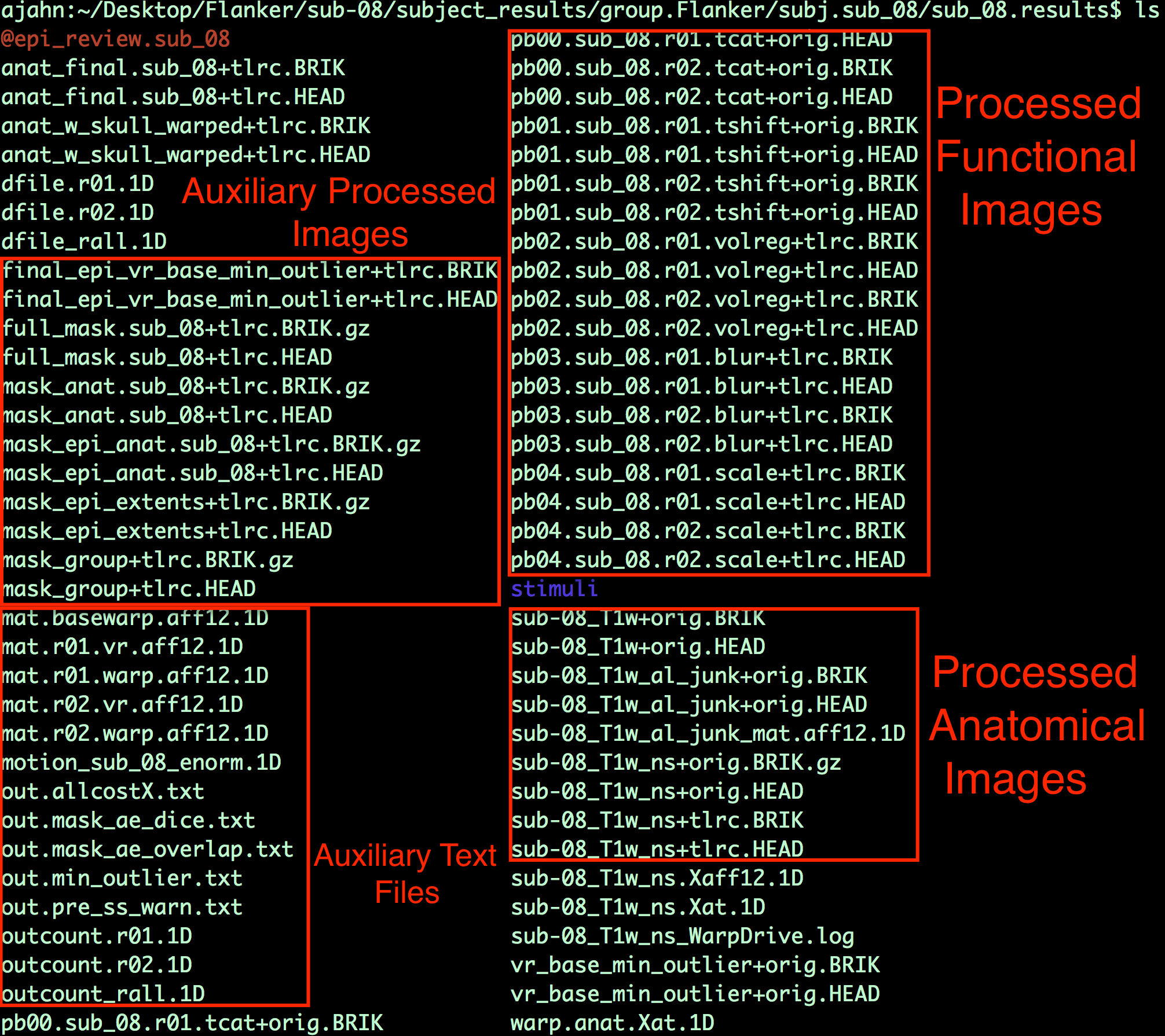
2).查看已处理的功能像数据
a.查看时间层校正结果
在熟悉预处理数据目录中的内容后,键入afni打开 AFNI 图形用户界面。单击"Underlay"按钮,左键单击文件pb00.sub_08.r01.tcat;然后单击轴向、矢状或冠状视图旁边的 “Graph"按钮,查看时间序列。由于在将数据上传到OpenNeuro之前已经丢弃了初始体积,因此所有时间点的相对强度都是相同的。(事实上,本来就不需要3dTcat预处理步骤;不过,除了占用更多电脑内存之外,留着它也没什么不好)。
同样,pb01图像应该与pb00图像相同。如果检查预处理的输出文本,就会看到3dTshift时打印的一条信息,其中指出数据集"已经在时间上对齐(already aligned in time)",而且 “输出数据集只是输入数据集的副本(output dataset is just a copy of the input dataset)"。至此,这些文件与原始功能数据基本相同。你可以省略3dTcat和3dTshift这两个预处理步骤,重新分析这些数据,结果也是一样的。不过现在,请查看每次运行的这两个处理输出,以确保它们看起来确实相同,而且没有明显的伪影。
底层菜单有两列: 左列是文件名,右列是文件的标题信息。“epan"表示该文件是回声平面图像(即功能图像),而 “anat"表示该文件是解剖图像。(在 “epan"字符串旁边,“3D+t:146"表示这是一个三维图像,外加一个时间维度,有 146 个体积或时间点。
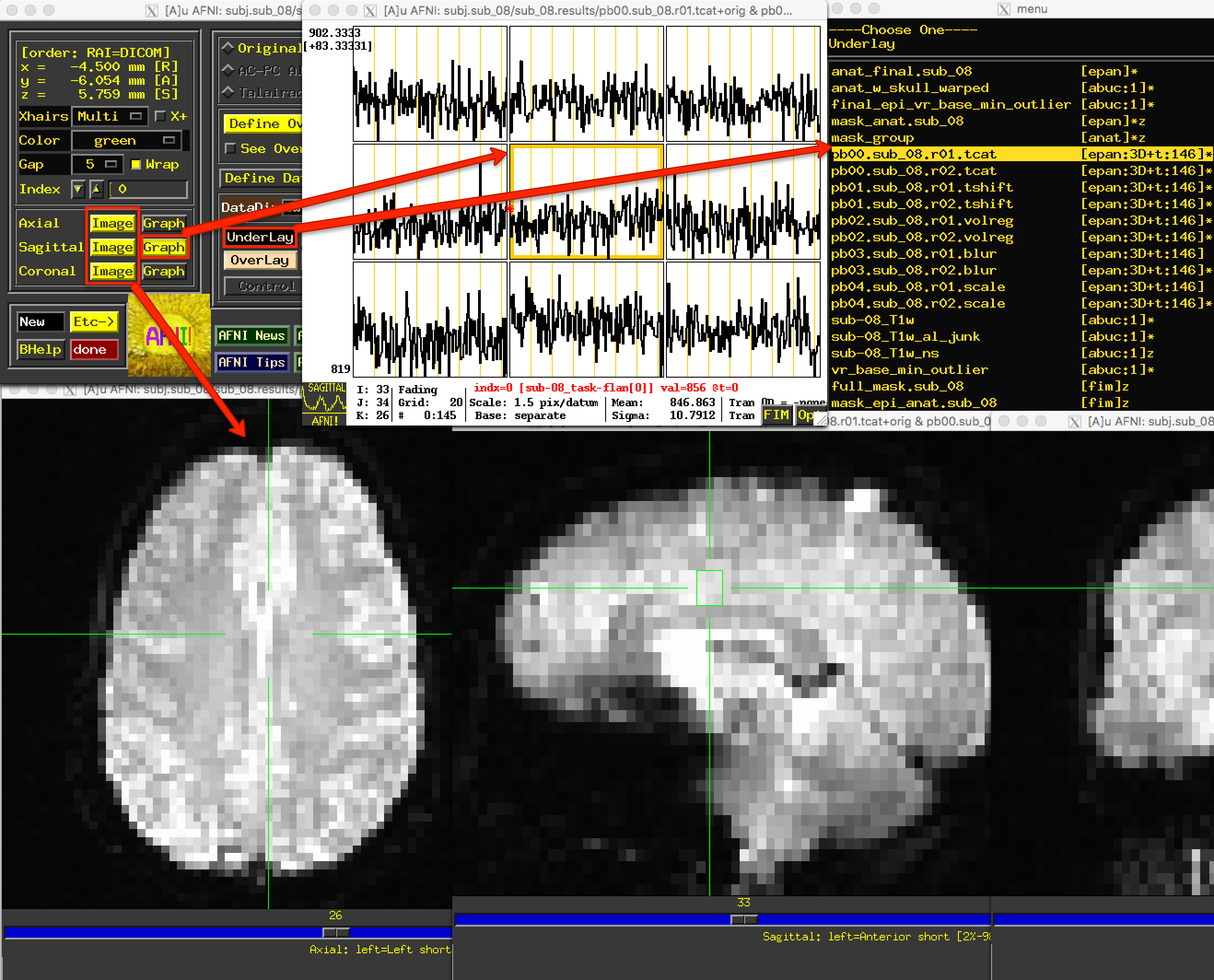
b.查看对齐与配准结果
下一个要查看的文件是pb02 “volreg"文件,这些文件已经过
- 运动校正–也就是说,每次运行的时间序列中的每个容积都已与参考容积对齐;
- 与解剖图像进行了共配准;3.
- 与标准化空间进行了扭曲(warp)处理,在本例中就是 MNI152 模板。
如果点击pb02图像,会发现视图发生了变化。AFNI GUI 有一部分包含 “Original View”、“AC-PC Aligned"和 “Talairach View"字符串。在该图像中,“Talairach View"单选按钮被高亮显示,表明这些图像已被归一化(normalized)。当查看其他被试的此处理块时,图像的基本形状和轮廓看起来几乎是一样的,因为它们都是按照相同的模板扭曲的。同样,请在几个不同的位置检查图像和时间序列,以确保没有明显的伪影。
在AFNI中,+tlrc扩展名(和 “Talairach View”)仅表示图像已被归一化。但这并不意味着图像一定是在 Talairach 空间中;出于传统目的(即为了确保代码在新版本中仍然有效),Talairach 标签被保留了下来。我们可以在图像上使用3dinfo命令,找到 “Template Space(模板空间)“字段,检查图像被扭曲到哪个空间–有三种可能:“ORIG”(即未被扭曲)、“TLRC”(归一化为 Talairach 空间)和 “MNI”(归一化为 MNI 空间)。
c.查看平滑结果 接下来的预处理步骤是平滑,它将附近体素的信号平均到一起,以增强任何存在的信号,并消除噪音。这些图像看起来会更模糊,这是应用于数据的平滑核大小的函数;在本例中,4毫米的平滑核会使数据略微模糊,但模糊程度不大。如下图所示,查看图像以确保模糊看起来合理。
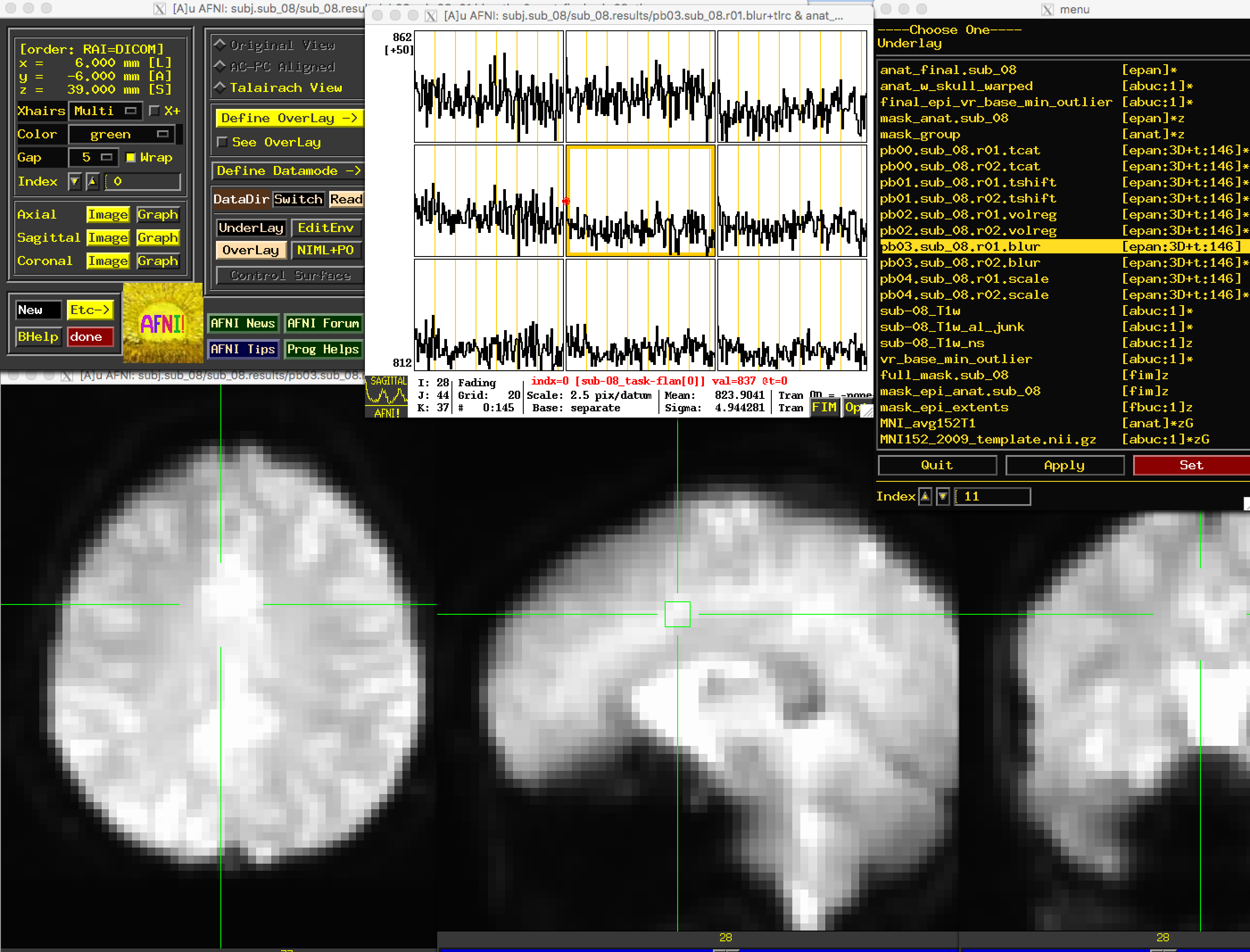
打开 “Graph"窗口,从 “volreg"图像切换到 “blur"图像时,确保十字准线位于同一体素上。你注意到时间序列有什么变化?有什么明显的变化吗?你如何描述这种变化,为什么你认为它发生了这种变化?
d.查看缩放结果 最后一个预处理步骤是生成缩放图像,其中每个体素的平均信号强度为100。这样,我们就可以将相对于平均值的任何变化指定为信号变化的百分比;也就是说,101的值可以解释为1%的信号变化。
由于图像灰度在脑部体素中更为均匀,而脑部以外的信号变化较大,因此这些图像的解剖学清晰度将低于之前的图像。不过,我们仍然可以看到大脑的轮廓,而且大脑体素的时间序列值应该都接近100:
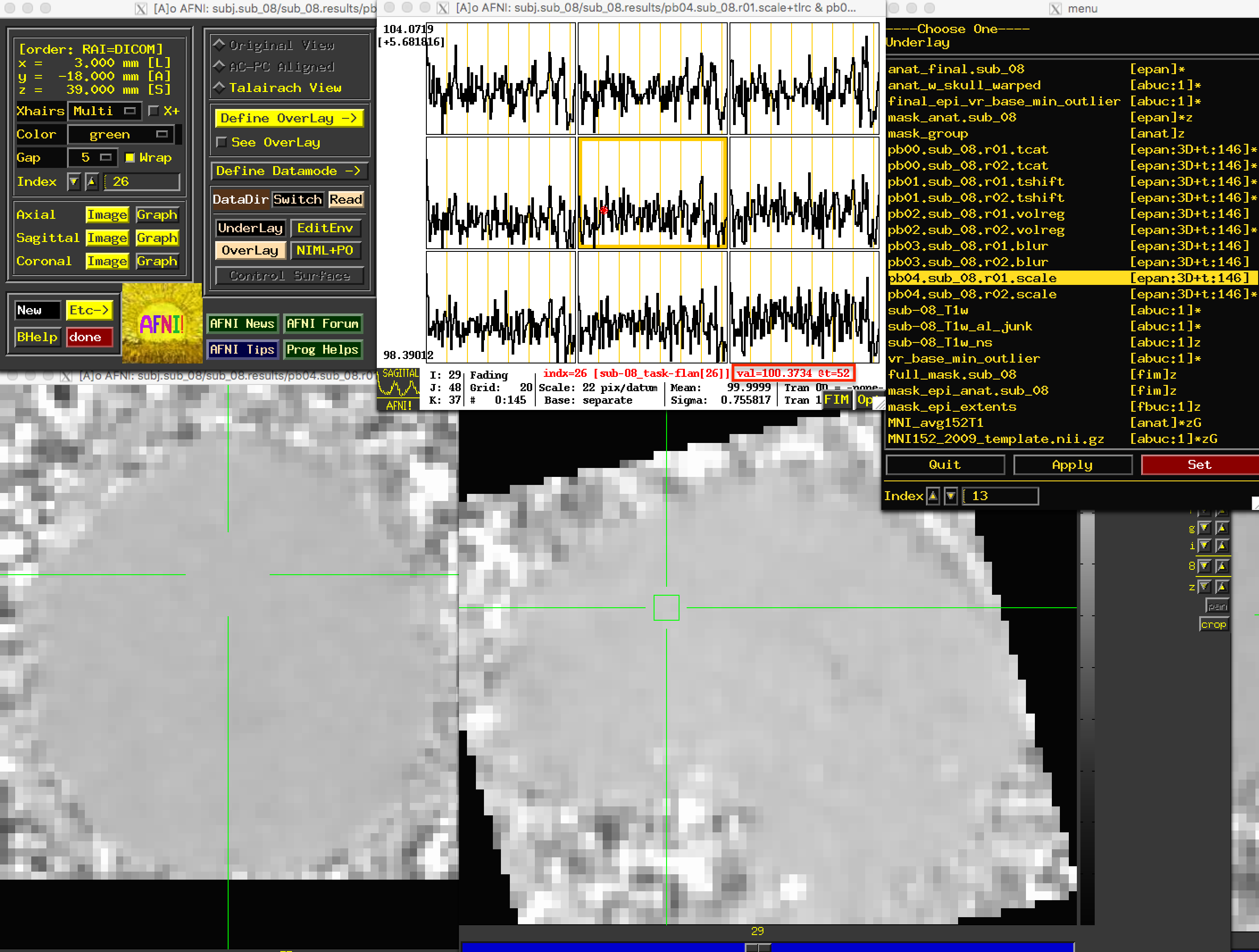 )
)
e.查看掩码结果 由于我们只对覆盖大脑的体素感兴趣,因此我们创建了一个掩码,用来排除任何非大脑体素。掩码将是二进制的:确定在头骨内的体素为1,头骨外为0。(还可以创建更严格的掩码,将脑脊液甚至白质排除在外,但我们在这里不考虑这些)。
有两种掩膜可供选择:full_mask 和mask_group。full_mask是所有单个功能图像掩膜的组合,这些掩膜已根据信号强度确定属于大脑。信号强度非常低的体素不被认为是大脑体素。正如你在full_mask中看到的那样,这也排除了眶额区的体素,而眶额区因容易出现信号丢失而臭名昭著:
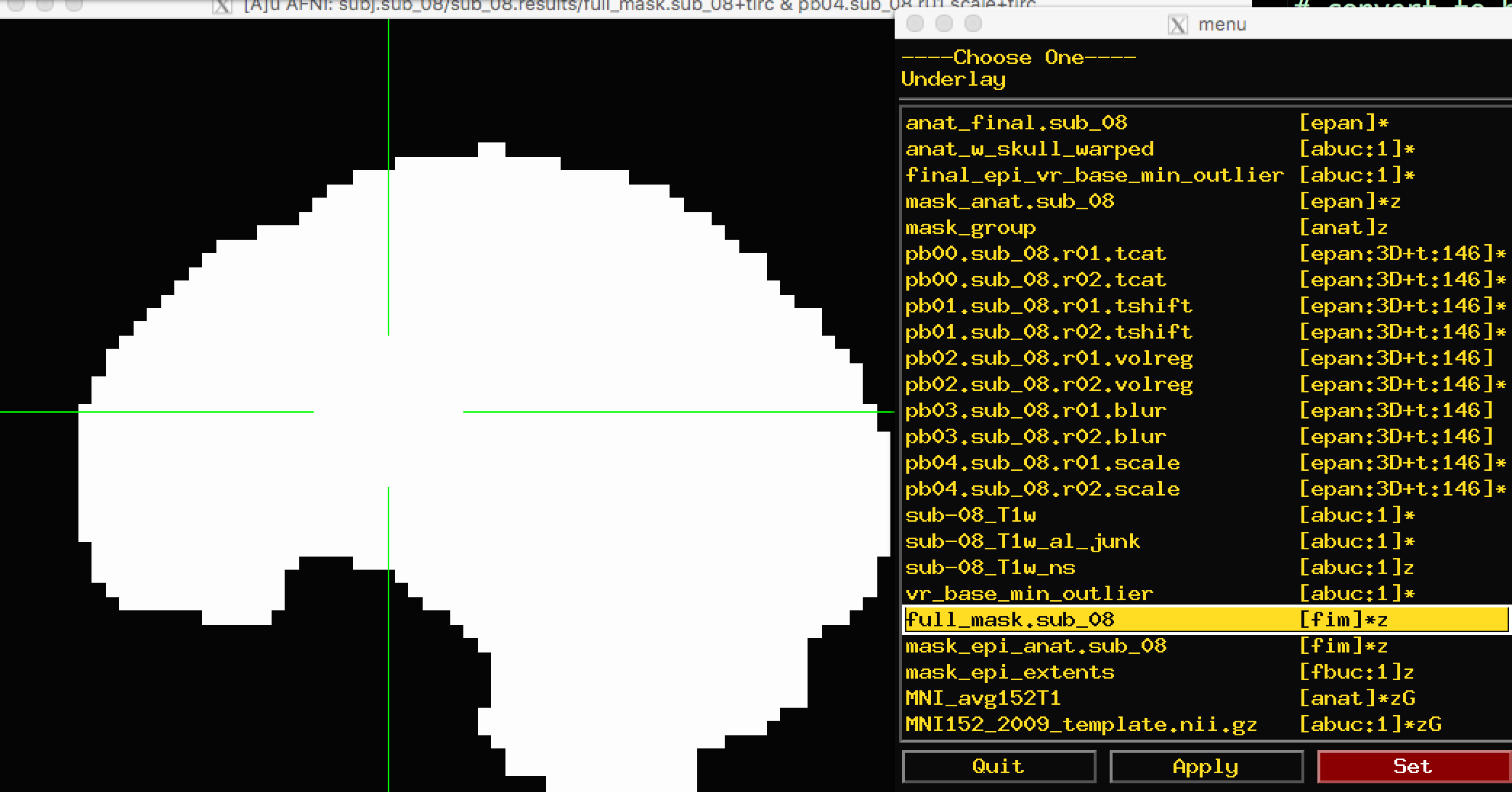
另一个掩码(mask_group)是一个更宽松的掩码,它已被放大,以更紧密地匹配您所扭曲的模板–在本例中,是MNI152大脑:
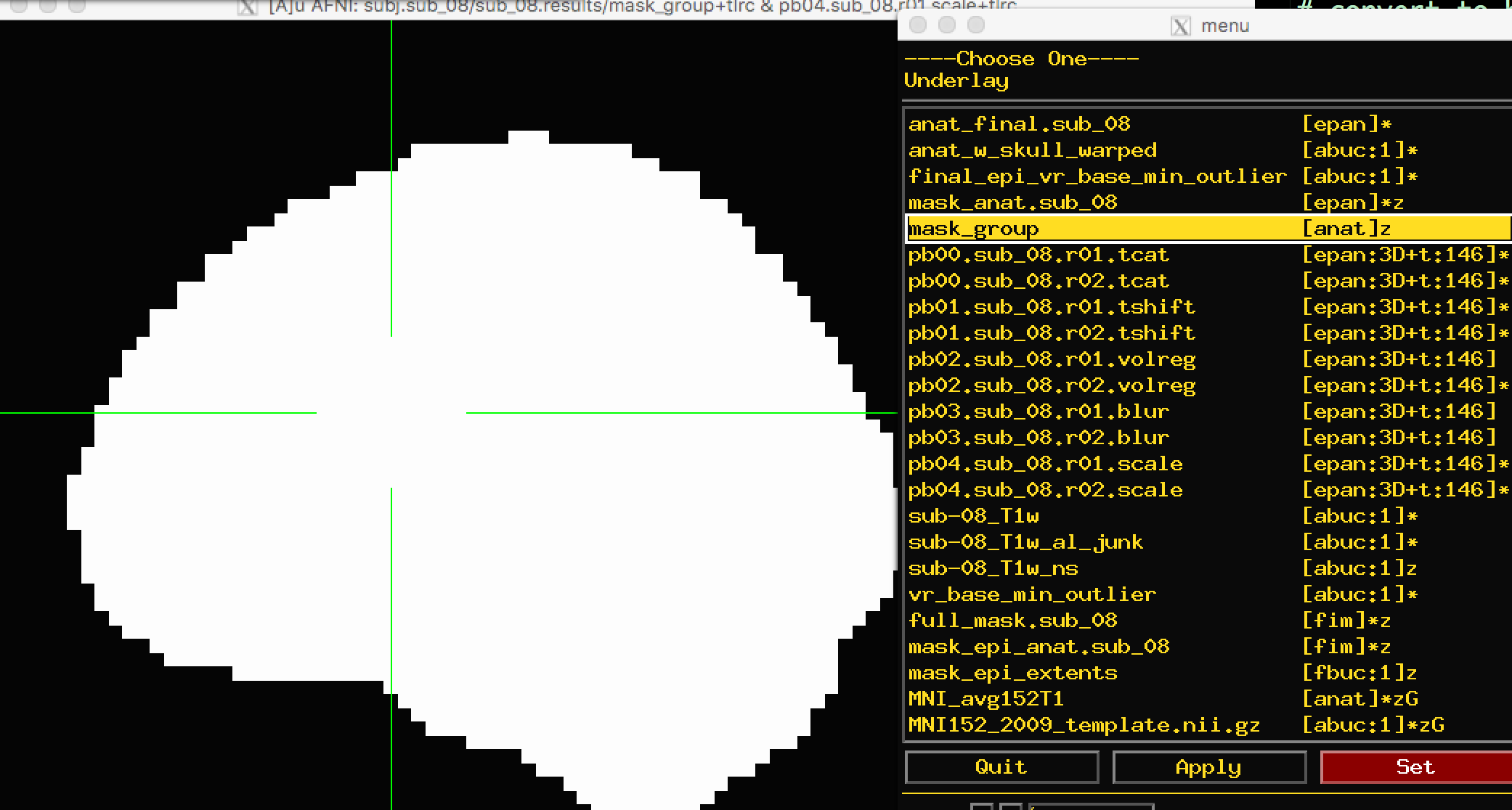
关于掩模图像的时间序列,你注意到什么?单击掩码内部和外部。这些数值有意义吗?
3).查看已处理的结构像数据 在查看解剖预处理的结果时,我们要确保颅骨去除后的图像看起来合理,而且图像的归一化处理得当。
首先,打开图像anat_w_skull_warped。如果已将MNI152图像复制到aglobal目录中,请将其作为叠加图像加载。(也可以通过终端键入:cp ~/abin/MNI_avg152T1+tlrc* .将其复制到当前目录)。我们可能会注意到,虽然矢状视图看起来不错,但轴向和冠状视图看起来有点糟。特别是,图像看起来好像略微向右偏移。虽然归一化过程中存在一些变异是很常见的,而且解剖图和模板永远不会完全匹配,但这已经超出了我们愿意扩大归一化误差的范围。
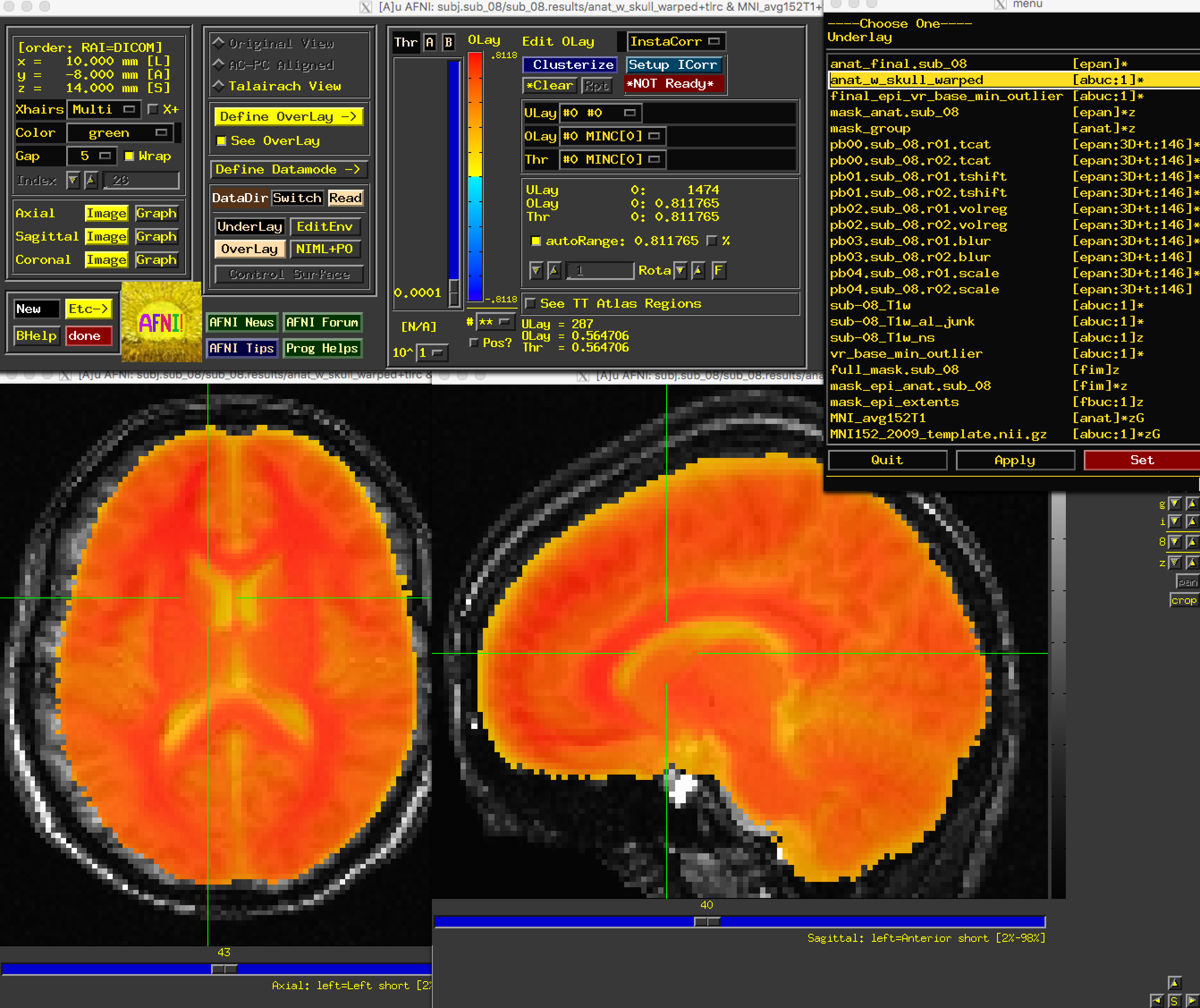
应该指出的是,anat_w_skull_warped图像是对原始解剖图像进行扭曲处理的结果。扭曲本身是通过将剥离头骨的解剖图像归一化为模板而计算出来的。如果归一化出现偏差,就会传播到其他图像上。要检查这一点,请作为底层(underlay)载入anat_final图像:
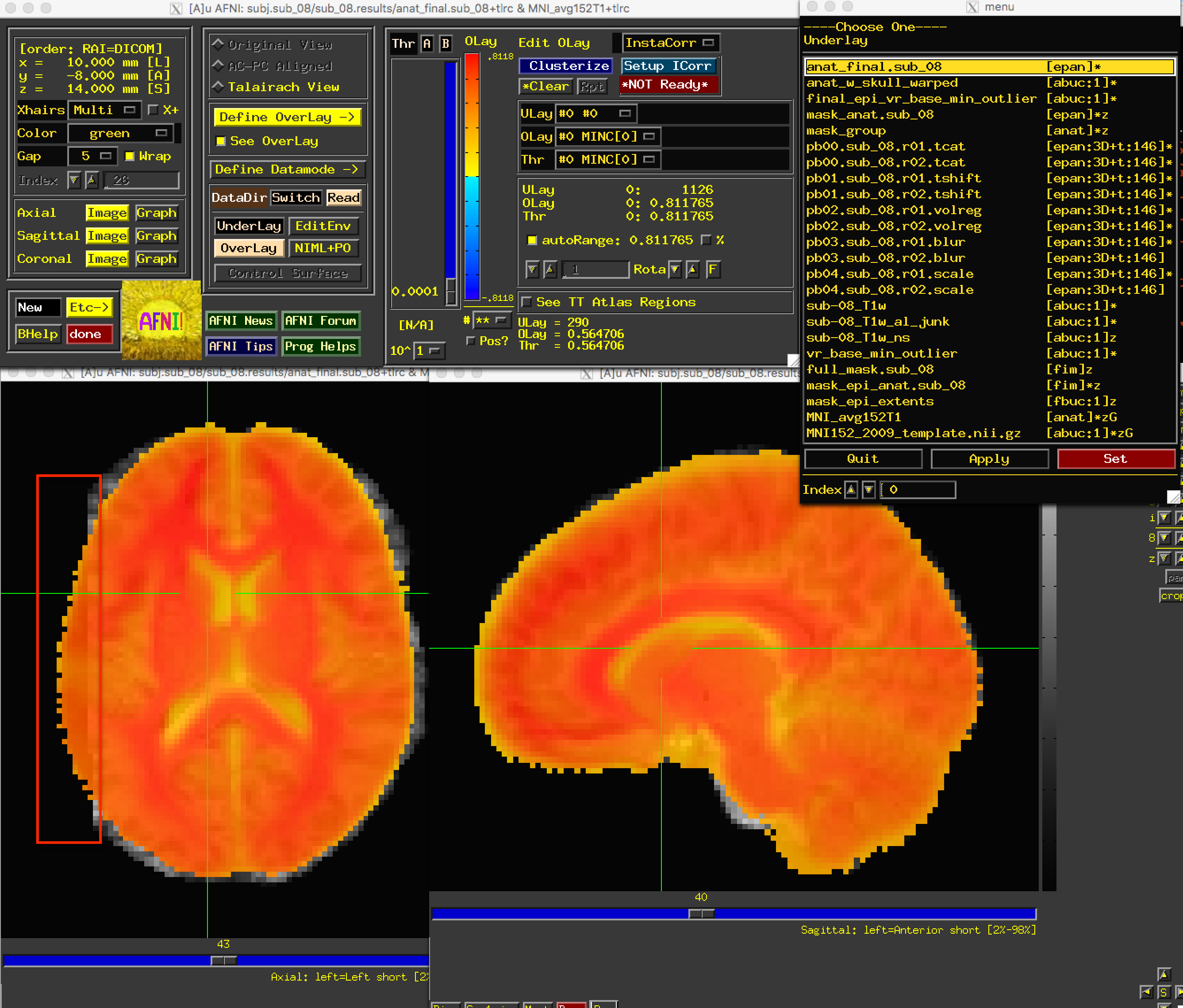 我们找到了错误的根源: 左侧大脑的一部分在正常化过程中被删除了。但我们该如何修复呢?
我们找到了错误的根源: 左侧大脑的一部分在正常化过程中被删除了。但我们该如何修复呢?
在预处理图像中发现错误时,应检查预处理脚本的输出。如果从uber_subject.py图形用户界面启动脚本,输出结果将打印到 “Processing Command"窗口;文本副本也将存储在名为:output.proc.<subjID 的文件中,该文件位于预处理数据的上一级目录。
该文本将包含 “Warnings"和 “Errors”。错误表示文件丢失或命令无法成功运行。通常,脚本会在遇到错误后退出。而警告则指出可能存在的问题。例如,我们在时间层校正时收到的"数据集已在时间上对齐"通知就是一个警告。
另一个警告与我们当前的问题有关,发生在归一化步骤中。这个警告出现在输出的中段,即@auto_tlrc命令之后:

显然,解剖图像和模板图像的中心相距甚远。输出结果显示,“如果部分原始解剖图被裁剪(if parts of the orignal anatomy gets cropped [sic])"(这正是我们目前的问题所在),“请尝试在@auto_tlrc命令中添加选项-init_xform AUTO_CENTER (try adding option -init_xform AUTO_CENTER to your @auto_tlrc command)"。我们可以这样做:导航到预处理目录之上的一个目录(cd ..),删除预处理目录(rm -r sub_08.results),然后编辑proc.sub_08文件,在@auto_tlrc命令后加入-init_xform AUTO_CENTER字符串,这应该是 proc 文件中的第 119 行:
@auto_tlrc -base MNI_avg152T1+tlrc -input sub-08_T1w_ns+orig -no_ss -init_xform AUTO_CENTER
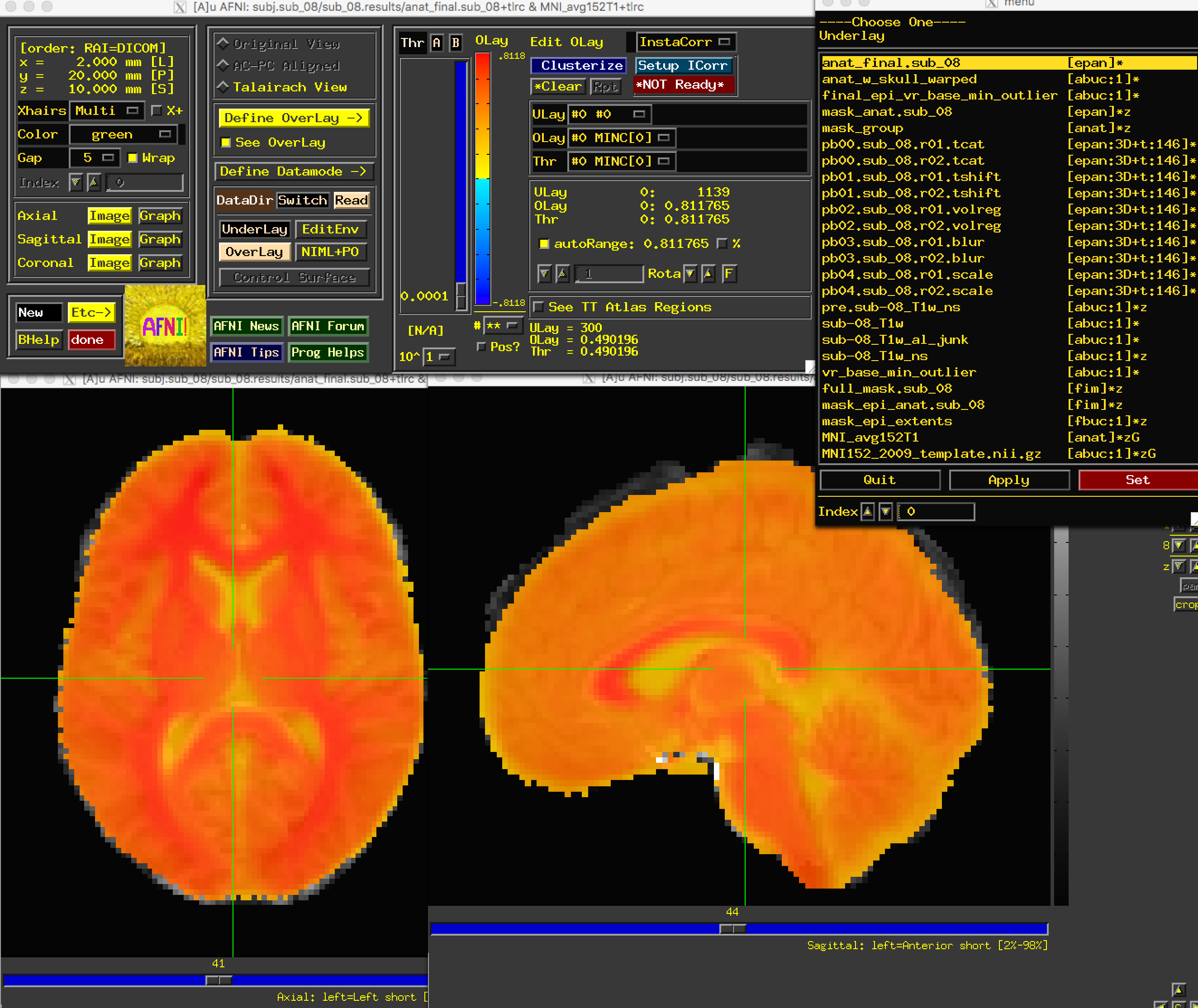
保存文件,然后输入tcsh proc.sub_08重新运行。等待几分钟让它完成,然后导航到预处理目录,加载与之前相同的图像集。现在你应该看到问题已经解决了。
练习
- 重新运行分析,使用10毫米的平滑核。预处理步骤的哪些部分会受到影响?在运行脚本之前,想一想输出结果会是什么样子。
- 在归一化解剖图像上叠加完整掩膜图像(
full_mask)和掩膜组图像(mask_group)(或将它们叠加到经压缩后的模板上,即MNI152图像)。注意到掩码之间有什么不同?掩膜的覆盖范围在哪里差异最大?为什么?